 |
 |
AbstractThe revolutionary development of artificial intelligence (AI) such as machine learning and deep learning have been one of the most important technology in many parts of industry, and also enhance huge changes in health care. The big data obtained from electrical medical records and digitalized images accelerated the application of AI technologies in medical fields. Machine learning techniques can deal with the complexity of big data which is difficult to apply traditional statistics. Recently, the deep learning techniques including convolutional neural network have been considered as a promising machine learning technique in medical imaging applications. In the era of precision medicine, otolaryngologists need to understand the potentialities, pitfalls and limitations of AI technology, and try to find opportunities to collaborate with data scientists. This article briefly introduce the basic concepts of machine learning and its techniques, and reviewed the current works on machine learning applications in the field of otolaryngology and rhinology.
ņä£ ļĪĀņØĖĻ│Ąņ¦ĆļŖź(artificial intelligence)ņØĆ Ļ│╝ĒĢÖ ĻĖ░ņłĀņØ┤ ļ░£ņĀäĒĢśļ®┤ņä£ ĻĘĖ Ļ░£ļģÉļÅä ĒĢ©Ļ╗ś ļ│ĆĒĢśĻ│Ā ņ׳ĻĖ░ ļĢīļ¼ĖņŚÉ ņĀĢņØśļź╝ ļé┤ļ”¼ĻĖ░ ņēĮņ¦Ć ņĢŖņ¦Ćļ¦ī, Ļ░äļŗ©Ē׳ ņäżļ¬ģĒĢśļ®┤ ņØĖĻ░äņØś ņ¦ĆļŖźņØä ļ¬©ļ░®ĒĢśņŚ¼ ņé¼ļ×īņØ┤ ĒĢśļŖö Ļ▓āĻ│╝ Ļ░ÖņØ┤ ļ│Ąņ×ĪĒĢ£ ņØ╝ņØä ĒĢĀ ņłś ņ׳ļŖö ĻĖ░Ļ│äļź╝ ļ¦īļō£ļŖö Ļ▓āņØä ļ¦ÉĒĢ£ļŗż[1]. ņ╗┤Ēō©Ēä░ ĒĢśļō£ņø©ņ¢┤ņÖĆ Ļ│ĄĒĢÖ ļō▒ Ļ┤ĆļĀ© ļČäņĢ╝Ļ░Ć ļ╣Āļź┤Ļ▓ī ļ░£ņĀäĒĢśĻ│Ā ņŚäņ▓Łļé£ ņ¢æņØś ļŹ░ņØ┤Ēä░ļź╝ ļ╣ĀļźĖ ņŗ£Ļ░ä ļé┤ņŚÉ ĒÜ©Ļ│╝ņĀüņ£╝ļĪ£ ļŗżļŻ░ ņłś ņ׳Ļ▓ī ļÉśļ®┤ņä£ ņØĖĻ│Ąņ¦ĆļŖź ĻĖ░ņłĀņØĆ ņØ┤ņĀ£ ņåīņäż ņåŹņŚÉ ļō▒ņןĒĢśļŖö ĒÖśņāüņØ┤ ņĢäļŗłļØ╝ ĒśäņŗżņØ┤ ļÉśņ¢┤Ļ░ĆĻ│Ā ņ׳ļŗż. ņĄ£ĻĘ╝ņŚÉļŖö ņŚ¼ļ¤¼ ņØĖĻ│Ąņ¦ĆļŖź ĻĖ░ņłĀ ņżæņŚÉņä£ļÅä ņØĖĻ░äņØ┤ ļ¦īļōĀ ĒöäļĪ£ĻĘĖļש ņŚåņØ┤ ņ╗┤Ēō©Ēä░Ļ░Ć ņŖżņŖżļĪ£ ļŹ░ņØ┤Ēä░ļź╝ ĒĢÖņŖĄĒĢśņŚ¼ ĻĘĖ ņØśļ»Ėļź╝ ĒĢ┤ņäØĒĢ┤ ļé┤ļŖö ļ©ĖņŗĀļ¤¼ļŗØ(machine learning)Ļ│╝ ļöźļ¤¼ļŗØ(deep learning) ĻĖ░ņłĀņØ┤ ņŗżņĀ£ ļ¼ĖņĀ£ ĒĢ┤Ļ▓░ņŚÉ ņĀüņÜ®ļÉśĻĖ░ ņŗ£ņ×æĒĢśļ®┤ņä£ ņØĖĻ│Ąņ¦ĆļŖź ņä▒ļŖźņØś ļ╣äņĢĮņĀüņØĖ ļ░£ņĀäņØä Ļ░ĆņĀĖņÖöļŗż. ņØśļŻī ļČäņĢ╝ņŚÉņä£ļŖö ņĀäņ×ÉņØśļ¼┤ĻĖ░ļĪØ(electrical medical record)ņØ┤ļéś ņØśļŻī ņśüņāü ņĀäņåĪ ņŗ£ņŖżĒģ£(picture archiving and communication system) ļō▒ņØä ĒåĄĒĢ┤ ĻĖ░ņĪ┤ņŚÉļŖö ļ│╝ ņłś ņŚåņŚłļŹś ļ╣ģļŹ░ņØ┤Ēä░ļź╝ ņČĢņĀüĒĢĀ ņłś ņ׳Ļ▓ī ļÉśĻ│Ā, ņØ┤ļ¤¼ĒĢ£ ļ╣ģļŹ░ņØ┤Ēä░ļź╝ ņØ┤ņÜ®ĒĢ£ ņØĖĻ│Ąņ¦ĆļŖź ļČäņĢ╝ņØś ĻĖ░ņłĀņØ┤ Ļ┤æļ▓öņ£äĒĢśĻ▓ī ļÅäņ×ģļÉśņ¢┤ ņØ╝ļČĆļŖö ņ×äņāüņŚÉņä£ ņĀüņÜ®ļÉśĻĖ░ ņŗ£ņ×æĒ¢łļŗż. ņØ┤ļ¤¼ĒĢ£ ĒØÉļ”äņØĆ Ē¢źĒøä ņØśļŻīĻ│ä ņĀäņ▓┤ņŚÉļÅä Ēü░ ļ│ĆĒÖöļź╝ Ļ░ĆņĀĖņś¼ Ļ▓āņ£╝ļĪ£ ļ│┤ņØĖļŗż.
ļ│Ė ņóģņäżņŚÉņä£ļŖö ļ©ĖņŗĀļ¤¼ļŗØĻ│╝ ļöźļ¤¼ļŗØņØś ĻĖ░ļ│Ė Ļ░£ļģÉļōżĻ│╝ ļīĆĒæ£ņĀüņØĖ ņĢīĻ│Āļ”¼ņ”ś ļ¬©ļŹĖļōżņØä Ļ░äļץĒ׳ ņäżļ¬ģĒĢśĻ│Ā, ņĄ£ĻĘ╝ ņØ┤ļ╣äņØĖĒøäĻ│╝ņÖĆ ļ╣äĻ│╝ ņśüņŚŁņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØä ņĀüņÜ®ĒĢ£ ņŚ░ĻĄ¼ļōżņØä ņåīĻ░£ĒĢśĻ│Āņ×É ĒĢ£ļŗż.
ļ©ĖņŗĀļ¤¼ļŗØ(Machine Learning)ņØĖĻ│Ąņ¦ĆļŖźņØĆ ĒżĻ┤äņĀüņ£╝ļĪ£ ŌĆśņØĖĻ░äņØś ņ¦ĆļŖźņØä ņØĖĻ│ĄņĀüņ£╝ļĪ£ ļ¬©ņé¼ĒĢśļŖö Ļ▓āŌĆÖņØä ņØśļ»ĖĒĢśļ®░, ņŚ¼ļ¤¼ ņØĖĻ│Ąņ¦ĆļŖź ļ░®ļ▓Ģ ņżæ Ēü¼Ļ▓ī ŌĆśĻĘ£ņ╣Ö ĻĖ░ļ░śŌĆÖņ£╝ļĪ£ ņé¼ņĀäņŚÉ ĻĘ£ņ╣ÖņØä ņĀĢĒĢ┤ņä£ ĒīÉļŗ©, ņČöļĪĀĒĢśļŖö ļ░®ļ▓ĢĻ│╝ ļŹ░ņØ┤Ēä░ļź╝ ĒåĄĒĢ┤ņä£ ļ¬©ĒśĢņØä ĒĢÖņŖĄĒĢśĻ▓ī ĒĢśļŖö ŌĆśļ©ĖņŗĀļ¤¼ļŗØ(ĻĖ░Ļ│äĒĢÖņŖĄ)ŌĆÖ ļČäņĢ╝ļĪ£ ļéśļłī ņłś ņ׳ļŗż. ĻĘ£ņ╣ÖņØä ĻĖ░ļ░śņ£╝ļĪ£ ĒĢśļŖö ļ░®ļ▓ĢņØĆ ņØĖĻ░äņØ┤ ļ»Ėļ”¼ ņĀĢĒĢ┤ļæö ĻĘ£ņ╣ÖņØä ĒåĄĒĢ┤ņä£ ĒīÉļŗ©ņØä ĒĢśĻ▓ī ļÉśņ¦Ćļ¦ī, ļ©ĖņŗĀļ¤¼ļŗØņØĆ ļŹ░ņØ┤Ēä░ļź╝ ĒĢÖņŖĄ ņĢīĻ│Āļ”¼ņ”śņŚÉ ņ¦æņ¢┤ļäŻņ¢┤ņä£ ĻĘĖļ¤¼ĒĢ£ ĻĘ£ņ╣ÖļōżņØä ņŖżņŖżļĪ£ ņ░ŠņØä ņłś ņ׳Ļ▓ī ļ¦īļō£ļŖö ņĢīĻ│Āļ”¼ņ”ś ļ░®ļ▓ĢņØ┤ļŗż. ļöźļ¤¼ļŗØņØĆ ņØ┤ļ¤¼ĒĢ£ ļ©ĖņŗĀļ¤¼ļŗØņØś ņŚ¼ļ¤¼ ļ░®ļ▓ĢļĪĀ ņżæņØś ĒĢśļéśļĪ£ ņØĖĻ│Ą ņŗĀĻ▓Įļ¦Ø(artificial neural network, ANN)ņØś ĒĢ£ ņóģļźśļØ╝Ļ│Ā ĒĢĀ ņłś ņ׳ļŗż(Fig. 1).
ļ©ĖņŗĀļ¤¼ļŗØņØĆ 1959ļģäņŚÉ ņ▓śņØī ņåīĻ░£ļÉĀ ņĀĢļÅäļĪ£ ņśżļלļÉ£ Ļ░£ļģÉņØ┤ņ¦Ćļ¦ī, ņĄ£ĻĘ╝ņŚÉ ļōżņ¢┤ņä£ņĢ╝ ņŚ¼ļ¤¼ ļČäņĢ╝ņŚÉņä£ ļäÉļ”¼ ņØ┤ņÜ®ļÉśĻ│Ā ņ׳ļŖöļŹ░, ņØ┤ļŖö Ļ│ĀņåŹ ņØĖĒä░ļäĘņØś ļ│┤ĒÄĖĒÖö ļ░Å ņ╗┤Ēō©Ēä░ ĒĢśļō£ņø©ņ¢┤ņØś ļ░£ņĀä, ĻĘĖļ”¼Ļ│Ā ļ¼┤ņŚćļ│┤ļŗż ļ╣ģļŹ░ņØ┤Ēä░ņØś ļō▒ņןņØ┤ Ļ░Ćņן Ēü░ ņØ┤ņ£ĀļØ╝Ļ│Ā ĒĢĀ ņłś ņ׳ļŗż. ļ©ĖņŗĀļ¤¼ļŗØņØś ĒĢÖņŖĄņŚÉļŖö ĻĖ░ļ│ĖņĀüņ£╝ļĪ£ ļ¦ÄņØĆ ņ¢æņØś ļŹ░ņØ┤Ēä░Ļ░Ć ĒĢäņÜöĒĢśĻ│Ā, ļŹ░ņØ┤Ēä░ņØś ņ¢æņŚÉ ļö░ļØ╝ ļ©ĖņŗĀļ¤¼ļŗØņØś ņä▒ļŖźņØ┤ ņóīņÜ░ļÉśļ»ĆļĪ£ ņČ®ļČäĒĢ£ ļŹ░ņØ┤Ēä░ņØś ĒÖĢļ│┤ļŖö ņØĖĻ│Ąņ¦ĆļŖź Ļ░£ļ░£ņŚÉņä£ Ļ░Ćņן ĻĖ░ļ│ĖņĀüņØĖ ņÜöņåīļØ╝Ļ│Ā ĒĢĀ ņłś ņ׳ļŗż. ļŹ░ņØ┤Ēä░ņØś ņ¦ł(quality) ņŚŁņŗ£ ļ©ĖņŗĀļ¤¼ļŗØņŚÉņä£ ņżæņÜöĒĢ£ ņÜöņåīņØ┤ļŗż. ĒĢÖņŖĄņŚÉ ĒĢäņÜöĒĢ£ ņĪ░Ļ▒┤ņØä Ļ░¢ņČöņ¦Ć ļ¬╗ĒĢ£ ņśżļźśĻ░Ć ņ׳ļŖö ļŹ░ņØ┤Ēä░ļŖö ļ©ĖņŗĀļ¤¼ļŗØņØś ņä▒ļŖźņØä ņĀĆĒĢśņŗ£ĒéżĻ│Ā Ļ▓░Ļ│╝ņŚÉ ņśüĒ¢źņØä ļü╝ņ╣Ā ņłś ņ׳ņ£╝ļ»ĆļĪ£, ĒĢÖņŖĄ ņØ┤ņĀäņŚÉ ļ░śļō£ņŗ£ ļŹ░ņØ┤Ēä░ņØś ņ¦łņØä ĒÅēĻ░ĆĒĢśļŖö Ļ│╝ņĀĢņØ┤ ņłśļ░śļÉśņ¢┤ņĢ╝ ĒĢ£ļŗż.
ļ©ĖņŗĀļ¤¼ļŗØĻ│╝ ĻĖ░ņĪ┤ ĒåĄĻ│äĒĢÖņØś ņ░©ņØ┤ļ©ĖņŗĀļ¤¼ļŗØĻ│╝ ĻĖ░ņĪ┤ņØś ņĀäĒåĄņĀüņØĖ ĒåĄĻ│äĒĢÖ(traditional statistics)ņØĆ ļŹ░ņØ┤Ēä░ļź╝ ĒåĄĒĢ┤ņä£ ļ¼ĖņĀ£ļź╝ ĒĢ┤Ļ▓░ĒĢ£ļŗżļŖö ļ®┤ņŚÉņä£ ņä£ļĪ£ ļ╣äņŖĘĒĢśļŗż. ņŗżņĀ£ļĪ£ ņäĀĒśĢ ĒÜīĻĘĆ(linear regression)ņÖĆ Ļ░ÖņØ┤ ļ©ĖņŗĀļ¤¼ļŗØņØś ļ¦ÄņØĆ ĻĖ░ļ▓ĢļōżņØ┤ ĒåĄĻ│äĒĢÖņØś ĻĖ░ļ▓ĢļōżĻ│╝ ņāüļŗ╣Ē׳ ņ£Āņé¼ĒĢśļŗż. ļŗżļ¦ī ļ¬®Ēæ£ņÖĆ ņĀäļץņŚÉņä£ ņä£ļĪ£ ņ░©ņØ┤Ļ░Ć ņ׳ļŗżĻ│Ā ĒĢĀ ņłś ņ׳ļŗż. ņĀäĒåĄņĀü ĒåĄĻ│äĒĢÖņØĆ ņØ╝ļ░śņĀüņ£╝ļĪ£ ņ×æņØĆ ļŹ░ņØ┤Ēä░ņģŗ(data set)ņØä ļŗżļŻ©ļ®░, Ļ▓░ļĪĀņŚÉ ņØ┤ļź┤ĻĖ░Ļ╣īņ¦Ć ņŚ¼ļ¤¼ ņØĖņ×Éļōż Ļ░äņØś ņāüĻ┤ĆĻ┤ĆĻ│äņŚÉ ļīĆĒĢ£ ņČöļĪĀ(inference)Ļ│╝ Ļ░ĆņĀĢ(assumption)ņØä ņżæņŗ£ĒĢ£ļŗż. ĒĢśņ¦Ćļ¦ī ļ©ĖņŗĀļ¤¼ļŗØņØĆ ļ╣ģļŹ░ņØ┤Ēä░ņŚÉ ļ│┤ļŗż ņĀüĒĢ®ĒĢśļ®░, ĻĖ░ņĪ┤ņØś ļŹ░ņØ┤Ēä░ļź╝ ļ░öĒāĢņ£╝ļĪ£ ņĢī ņłś ņŚåļŖö ļČĆļČäņŚÉ ļīĆĒĢ£ ņĀĢĒÖĢĒĢ£ ņśłņĖĪ(prediction)ņŚÉ ņ¦æņżæĒĢ£ļŗż[2]. ļæÉĻ▓ĮļČĆ ņĢöņØś ņāØņĪ┤ņ£©ņØä ņśłļĪ£ ļōżļ®┤, ĻĖ░ņĪ┤ņØś ĒåĄĻ│äĒĢÖņŚÉņä£ļŖö ņāØņĪ┤ņ£©ņŚÉ ņśüĒ¢źņØä ļ»Ėņ╣śļŖö ņŚ¼ļ¤¼ ņØĖņ×ÉļōżĻ│╝ņØś ņāüĻ┤ĆĻ┤ĆĻ│äļź╝ ļČäņäØĒĢśļŖö ļŹ░ ņ┤łņĀÉņØ┤ ļ¦×ņČ░ņĀĖ ņ׳ļŗżļ®┤, ļ©ĖņŗĀļ¤¼ļŗØņŚÉņä£ļŖö ĻĖ░ņĪ┤ņØś ļŹ░ņØ┤Ēä░ļź╝ ĒåĄĒĢ┤ņä£ ņāłļĪ£ņÜ┤ ĒÖśņ×ÉņØś ņāØņĪ┤ņ£©ņØä ņĀĢĒÖĢĒĢśĻ▓ī ņśłņĖĪĒĢśļŖö ļŹ░ ņ¦æņżæĒĢ£ļŗżĻ│Ā ĒĢĀ ņłś ņ׳ļŗż.
ļ©ĖņŗĀļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņśłņĖĪ ļ¬©ļŹĖļ¦üņØś ņ¦äĒ¢ēĻ│╝ņĀĢļ©ĖņŗĀļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņśłņĖĪ ļ¬©ļŹĖļ¦ü(predictive modeling)ņØĆ ņØ╝ļ░śņĀüņ£╝ļĪ£ ļŗżņØīĻ│╝ Ļ░ÖņØĆ Ļ│╝ņĀĢņØä Ļ▒░ņ╣śĻ▓ī ļÉ£ļŗż.
1) ļŹ░ņØ┤Ēä░ ņłśņ¦æ(data collection)
2) ļŹ░ņØ┤Ēä░ ņĀäņ▓śļ”¼(data preprocessing): ļŹ░ņØ┤Ēä░ļź╝ ņĀĢņĀ£ĒĢ┤ņä£ ļ©ĖņŗĀļ¤¼ļŗØ ļ¬©ļŹĖņØś ņ×ģļĀźņŚÉ ņĀüĒĢ®ĒĢ£ ĒśĢĒā£ļĪ£ ļ░öĻ┐öņŻ╝Ļ▒░ļéś, ļŹ░ņØ┤Ēä░ņØś ĻĖ░ņĪ┤ ņåŹņä▒(feature)ņØä ņĪ░ĒĢ®ĒĢ┤ņä£ ņāłļĪ£ņÜ┤ ņåŹņä▒ņØä ņāØņä▒
3) ĒāÉņāēņĀü ļŹ░ņØ┤Ēä░ ļČäņäØ(exploratory data analysis): ļŹ░ņØ┤Ēä░ņØś ĒŖ╣ņ¦ĢņØä ņ░ŠĻ│Ā, ņł©Ļ▓©ņ¦ä Ēī©Ēä┤ņØä ļ░£Ļ▓¼
4) ļ¬©ļŹĖ ņäĀĒāØ(model selection): ņŻ╝ņ¢┤ņ¦ä ļ¼ĖņĀ£ņÖĆ ļŹ░ņØ┤Ēä░ņŚÉ ļ¦×ļŖö ņĀüņĀłĒĢ£ ļ¬©ļŹĖĻ│╝ ņåŹņä▒ņØä ņäĀĒāØ
5) ĒÅēĻ░Ć ļ░Å ņĀüņÜ®(evaluation & application): ļ¦īļōżņ¢┤ņ¦ä ļ©ĖņŗĀļ¤¼ļŗØ ļ¬©ļŹĖņØś ņä▒ļŖźņØä ĒÅēĻ░ĆĒĢśĻ│Ā, ļ¬©ļŹĖņØä ĒÖ£ņÜ®ĒĢśņŚ¼ ņāłļĪ£ņÜ┤ ļŹ░ņØ┤Ēä░ņŚÉ ļīĆĒĢ£ ņśłņĖĪņØä ņŗżĒ¢ē
Ļ│ĀņĀäņĀüņØĖ ļ©ĖņŗĀļ¤¼ļŗØ ņĢīĻ│Āļ”¼ņ”śņØś ņóģļźśļ©ĖņŗĀļ¤¼ļŗØņØĆ ņ¦ĆļÅäĒĢÖņŖĄ, ļ╣äņ¦ĆļÅäĒĢÖņŖĄĻ│╝ Ļ░ĢĒÖöĒĢÖņŖĄņØś ņäĖ Ļ░Ćņ¦Ć ņóģļźśļĪ£ ļéśļłī ņłś ņ׳ļŗż. ņ¦ĆļÅäĒĢÖņŖĄ(supervised learning)ņØĆ ņ×ģļĀźĻ░Æ(input)Ļ│╝ ĒĢ©Ļ╗ś ņĀĢļŗĄĻ░Æ(label)ņØä ņŻ╝Ļ│Ā ĒĢÖņŖĄņØä ņŗ£ĒéżļŖö ļ░®ļ▓Ģņ£╝ļĪ£, ņĢīĻ│Āļ”¼ņ”śņØä ņŗżĒ¢ēĒĢśļ®┤ņä£ ņ¢╗ņ¢┤ņ¦ä Ļ▓░Ļ│╝ņÖĆ ņĀĢļŗĄĻ░ÆņØä ĒĢ©Ļ╗ś ļ╣äĻĄÉĒĢśļ®┤ņä£ ļ░śļ│ĄņĀüņ£╝ļĪ£ ĒøłļĀ©ĒĢ┤ ļéśĻ░ĆļŖö ļ░®ļ▓ĢņØ┤ļŗż. Ēśäņ×¼ ņØ┤ņÜ®ļÉśĻ│Ā ņ׳ļŖö ļŗżņłśņØś ļ©ĖņŗĀļ¤¼ļŗØ ņĢīĻ│Āļ”¼ņ”śņØ┤ ņŚ¼ĻĖ░ņŚÉ ĒĢ┤ļŗ╣ĒĢśļ®░, ļČäļźś(classification)ņÖĆ ĒÜīĻĘĆ(regression) ļō▒ņØś ļ¼ĖņĀ£ļź╝ ĒĢ┤Ļ▓░ĒĢśļŖö ļŹ░ ņØ┤ņÜ®ļÉ£ļŗż. ņØ┤ņŚÉ ļ░śĒĢ┤ ļ╣äņ¦ĆļÅäĒĢÖņŖĄ(unsupervised learning)ņØĆ ņĀĢļŗĄĻ░ÆņØ┤ ņŚåņØ┤ ņ×ģļĀźĻ░Æļ¦īņØä ņØ┤ņÜ®ĒĢśņŚ¼ ĒĢÖņŖĄņØä ņŗ£ĒéżĻ▓ī ļÉśļ®░, ĒĢśņ£ä ļ▓öņŻ╝ļĪ£ļŖö Ēü┤ļ¤¼ņŖżĒä░ļ¦ü(clustering), ņ░©ņøÉ ņČĢņåī(dimensionality reduction) ļō▒ņØ┤ ņ׳ļŗż. ļ¦łņ¦Ćļ¦ēņ£╝ļĪ£ Ļ░ĢĒÖöĒĢÖņŖĄ(reinforcement learning)ņØĆ Ļ▓░Ļ│╝Ļ░Æ ļīĆņŗĀ ņ¢┤ļ¢ż ņ×æņŚģņØä ņל ņłśĒ¢ēĒ¢łņØä ļĢī ļ│┤ņāü(reward)ņØä ņŻ╝ļŖö ļ░®ļ▓Ģņ£╝ļĪ£ ĒĢÖņŖĄņØä ņŗ£Ēé©ļŗż(Fig. 2).
ļöźļ¤¼ļŗØ(Deep Learning) ņåīĻ░£ļöźļ¤¼ļŗØņØĆ ļ©ĖņŗĀļ¤¼ļŗØņØś ņŚ¼ļ¤¼ ļ░®ļ▓ĢļĪĀ ņżæņØś ĒĢśļéśļĪ£ ņØĖĻ│Ą ņŗĀĻ▓Įļ¦Ø(ANN)ņØ┤ ļ░£ņĀäļÉ£ Ļ░£ļģÉņØ┤ļØ╝Ļ│Ā ĒĢĀ ņłś ņ׳ļŗż. 1943ļģäņŚÉ ņ▓śņØī ņåīĻ░£ļÉ£ ņØĖĻ│Ą ņŗĀĻ▓Įļ¦ØņØĆ ļæÉļćīņØś ņŗĀĻ▓ĮņäĖĒż, ņ”ē ļē┤ļ¤░ņØ┤ ņŚ░Ļ▓░ļÉ£ ĒśĢĒā£ļź╝ ļ¬©ļ░®ĒĢ£ ļ¬©ļŹĖļĪ£, ņØĖĻ░äņØś ņŗĀĻ▓Į ĻĄ¼ņĪ░ļź╝ ļ│Ąņ×ĪĒĢ£ ņŖżņ£äņ╣śļōżņØ┤ ņŚ░Ļ▓░ļÉ£ ļäżĒŖĖņøīĒü¼ļĪ£ Ēæ£ĒśäĒĢĀ ņłś ņ׳ļŗżļŖö Ļ░£ļģÉņŚÉņä£ ņ£ĀļלļÉśņŚłļŗż[3]. ĻĘĖļ”¼Ļ│Ā 1958ļģä Rosenblatt [4]ņØĆ ĒŹ╝ņģēĒŖĖļĪĀ(perceptron)ņØ┤ļØ╝ļŖö ņäĀĒśĢļČäļźśĻĖ░ ļ¬©ļŹĖņØä ņĀ£ņĢłĒ¢łļŖöļŹ░, ņØ┤ļŖö ņ×ģļĀź(input)Ļ│╝ Ļ░Ćņżæņ╣ś(weight)ļōżņØś Ļ│▒ņØä ļ¬©ļæÉ ļŹöĒĢ£ ļÆż ĒÖ£ņä▒ĒÖö ĒĢ©ņłś(activation function)ļź╝ ņĀüņÜ®ĒĢ┤ņä£ ĻĘĖ Ļ░ÆņØ┤ 0ļ│┤ļŗż Ēü¼ļ®┤ 1, 0ļ│┤ļŗż ņ×æņ£╝ļ®┤ -1ņØä ņČ£ļĀźĒĢśļŖö ĻĄ¼ņĪ░ņśĆļŗż(Fig 3A). Ēśäņ×¼ ņé¼ņÜ®ĒĢśļŖö ļöźļ¤¼ļŗØ ļ¬©ļŹĖļÅä ĻĘ╝ļ│ĖņĀüņ£╝ļĪ£ ĒŹ╝ņģēĒŖĖļĪĀĻ│╝ Ļ░ÖņØĆ ĻĄ¼ņĪ░ļØ╝Ļ│Ā ĒĢĀ ņłś ņ׳ļŗż. ļŗżļ¦ī ņØ┤ ĻĄ¼ņĪ░ļź╝ ņŚ¼ļ¤¼ Ļ░£ņØś ļģĖļō£(node)ņÖĆ ļŗżņĖĄņØś ļĀłņØ┤ņ¢┤(layer)ļĪ£ ĒÖĢņןĒ¢łļŗżļŖö ņĀÉņŚÉņä£ ņ░©ņØ┤Ļ░Ć ņ׳ņØä ļ┐ÉņØ┤ļŗż. ĒĢśņ¦Ćļ¦ī ļŗ©ņł£ĒĢ£ ĒŹ╝ņģēĒŖĖļĪĀ ļ¦īņ£╝ļĪ£ļŖö ļ│Ąņ×ĪĒĢ£ ļ¼ĖņĀ£ļź╝ ĒÆĆ ņłś ņŚåņŚłĻĖ░ ļĢīļ¼ĖņŚÉ ņØĆļŗēĻ│äņĖĄ(hidden layer)ņØ┤ļØ╝ļŖö ņżæĻ░äņĖĄņØä ņČöĻ░ĆĒĢśņŚ¼ ĒĢ£Ļ│äļź╝ ĻĘ╣ļ│ĄĒĢ£ ļŗżņĖĄ ĒŹ╝ņģēĒŖĖļĪĀ(multilayer perceptron)ņØ┤ ņåīĻ░£ļÉśņŚłļŗż(Fig 3B). ņŚ¼ĻĖ░ņŚÉņä£ ĒŹ╝ņģēĒŖĖļĪĀņØä ņŚ¼ļ¤¼ ņĖĄņ£╝ļĪ£ ņīōņØäņłśļĪØ ĒĢÖņŖĄĒĢśĻĖ░ ņ¢┤ļĀżņÜ┤ ļ¼ĖņĀ£Ļ░Ć ņĀ£ĻĖ░ļÉśņŚłļŖöļŹ░, ņł£ļ░®Ē¢ź(feed forward) ņŚ░ņé░ Ēøä ņśłņĖĪ Ļ░ÆĻ│╝ ņĀĢļŗĄ ņé¼ņØ┤ņØś ņśżņ░©ļź╝ Ēøäļ░®(backward)ņ£╝ļĪ£ ļŗżņŗ£ ļ│┤ļé┤ņŻ╝ļ®┤ņä£ ĒĢÖņŖĄņŗ£ĒéżļŖö ļ░®ļ▓ĢņØĖ ņŚŁņĀäĒīī ņĢīĻ│Āļ”¼ņ”ś(backpropagtion algorithm)ņ£╝ļĪ£ ĒĢ┤Ļ▓░ļÉśņŚłļŗż[5]. ĒĢśņ¦Ćļ¦ī ļ│Ąņ×ĪĒĢ£ ļ¼ĖņĀ£ņØś ĒĢ┤Ļ▓░ņØä ņ£äĒĢ┤ Ļ╣ŖņØĆ ņĖĄņłśļź╝ ņīōņØä Ļ▓ĮņÜ░, ņŚŁņĀäĒīī ĒĢÖņŖĄĻ│╝ņĀĢņŚÉņä£ ļŹ░ņØ┤Ēä░Ļ░Ć ņé¼ļØ╝ņĀĖ ĒĢÖņŖĄņØ┤ ņל ļÉśņ¦Ć ņĢŖļŖö ĒśäņāüņØ┤ ļ░£ņāØĒĢśļ®┤ņä£ ņØĖĻ│Ą ņŗĀĻ▓Įļ¦ØņØĆ ņĀĢņ▓┤ĻĖ░ļź╝ Ļ▓¬ņŚłļŗż. 2000ļģäļīĆņŚÉ ļōżņ¢┤ņä£ļ®┤ņä£ Nair ļō▒[6]ņŚÉ ņØśĒĢ┤ ņé¼ņĀä ĒĢÖņŖĄ(pretraining)ņØ┤ļéś ReLUņÖĆ Ļ░ÖņØĆ ņāłļĪ£ņÜ┤ ĒÖ£ņä▒ĒĢ©ņłśņØś ņĀüņÜ®, ĻĘĖļ”¼Ļ│Ā ĒĢÖņŖĄ ļÅäņżæņŚÉ Ļ│ĀņØśļĪ£ ļŹ░ņØ┤Ēä░ļź╝ ļłäļØĮņŗ£ĒéżļŖö ļ░®ļ▓Ģ(dropout)[7] ļō▒ņ£╝ļĪ£ ņØ┤ļ¤¼ĒĢ£ ļ¼ĖņĀ£ļōżņØ┤ ĒĢ┤Ļ▓░ļÉśļ®┤ņä£ Ļ╣ŖņØĆ ņĖĄņØä Ļ░¢ļŖö ņŗĀĻ▓Įļ¦Ø ĒĢÖņŖĄ(deep neural network)ņØ┤ Ļ░ĆļŖźĒĢ┤ņĪīĻ│Ā, ņØ┤ĒøäņŚÉ ņØĖĻ│Ą ņŗĀĻ▓Įļ¦ØņØĆ ŌĆśļöźļ¤¼ļŗØ(deep learning)ŌĆÖņØ┤ļØ╝ļŖö ņāłļĪ£ņÜ┤ ņØ┤ļ”äņ£╝ļĪ£ ļČłļ”¼Ļ▓ī ļÉśņŚłļŗż. ļöźļ¤¼ļŗØņØ┤ ņĄ£ĻĘ╝ ļäÉļ”¼ ņ£ĀĒ¢ēĒĢśĻ▓ī ļÉ£ ņØ┤ņ£ĀņŚÉļŖö ĻĖ░ņĪ┤ ņØĖĻ│ĄņŗĀĻ▓Įļ¦ØņØś ĒĢ£Ļ│äļź╝ ĻĘ╣ļ│ĄĒĢ£ ņĢīĻ│Āļ”¼ņ”śņØś Ļ░£ļ░£Ļ│╝ ņŗĀĻ▓Įļ¦Ø ĒĢÖņŖĄņŚÉ ĒĢäņÜöĒĢ£ ļ¦ēļīĆĒĢ£ ĒĢÖņŖĄļŹ░ņØ┤Ēä░ņØś ņČĢņĀü, ĻĘĖļ”¼Ļ│Ā ņŗĀĻ▓Įļ¦Ø ĒĢÖņŖĄņŚÉ ņĀüĒĢ®ĒĢ£ ņ╗┤Ēō©Ēä░ ĒĢśļō£ņø©ņ¢┤ņØś ļ░£ņĀä ļō▒ņØä ļōż ņłś ņ׳ļŗż.
ļöźļ¤¼ļŗØĻ│╝ ļ©ĖņŗĀļ¤¼ļŗØ ļ╣äĻĄÉļöźļ¤¼ļŗØņØĆ ņŖżņŖżļĪ£ ļŹ░ņØ┤Ēä░ņØś ĒŖ╣ņä▒ņØä ņ░ŠņĢäļé┤ņ¢┤ ņŖżņŖżļĪ£ ĒĢÖņŖĄņØä ĒĢ£ļŗżļŖö ņĀÉņŚÉņä£ ĻĖ░ņĪ┤ņØś ļ©ĖņŗĀļ¤¼ļŗØĻ│╝ Ļ░Ćņן Ēü░ ņ░©ņØ┤Ļ░Ć ņ׳ļŗż. ĻĖ░ņĪ┤ņØś ļ©ĖņŗĀļ¤¼ļŗØ ĻĖ░ļ▓ĢņŚÉņä£ļŖö ņé¼ļ×īņØ┤ ņ¦üņĀæ ņČöņČ£ĒĢśĻ│Ā ļČäņäØĒĢ£ ļŹ░ņØ┤Ēä░ņØś ĒŖ╣ņä▒ņØä ĻĖ░ļ░śņ£╝ļĪ£ ĒĢÖņŖĄĒĢśņŚ¼ ļ¼ĖņĀ£ļź╝ ĒĢ┤Ļ▓░ĒĢśņśĆļŗż. ĒĢÖņŖĄ ĒŖ╣ņä▒ņØĆ ņłśĒ¢ēĒĢśļŖö ņ×æņŚģņŚÉ ļö░ļØ╝ ņóģļźśĻ░Ć ļ¦ÄĻ│Ā ņ¢┤ļ¢ĀĒĢ£ ĒŖ╣ņä▒ļōżņØä ņäĀĒāØĒĢśļŖöĻ░ĆņŚÉ ļö░ļØ╝ ļ¬©ļŹĖ ņä▒ļŖźņØś ĒĢ£Ļ│äĻ░Ć ņ׳ņŚłļŗż. ĒĢśņ¦Ćļ¦ī ļöźļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ĒĢÖņŖĄņŚÉņä£ļŖö ņŖżņŖżļĪ£ ņØ┤ļ¤¼ĒĢ£ ĒŖ╣ņä▒ļōżņØä ņ░ŠņĢäļé┤Ļ│Ā ĒŖ╣ņä▒ ĒĢÖņŖĄņØä ņłśĒ¢ēĒĢ£ļŗż. ņØ┤ļ░¢ņŚÉļÅä ļöźļ¤¼ļŗØĻ│╝ ĻĖ░ņĪ┤ņØś ļ©ĖņŗĀļ¤¼ļŗØ ĻĖ░ļ▓Ģ ņé¼ņØ┤ņŚÉļŖö ņŚ¼ļ¤¼ ņ░©ņØ┤ņĀÉļōżņØ┤ ņ׳ļŗż(Table 1).
ļīĆĒæ£ņĀüņØĖ ļöźļ¤¼ļŗØ ņĢīĻ│Āļ”¼ņ”ś: CNNĻ│╝ RNNņĄ£ĻĘ╝ ļäÉļ”¼ ņØ┤ņÜ®ļÉśļŖö ļöźļ¤¼ļŗØ ļ¬©ļŹĖņŚÉļŖö ĒĢ®ņä▒Ļ│▒ņŗĀĻ▓Įļ¦Ø(convolutional neural network, CNN)Ļ│╝ ņł£ĒÖś ņŗĀĻ▓Įļ¦Ø(recurrent neural network, RNN)ņØ┤ ņ׳ļŗż. ņŚ¼ĻĖ░ņŚÉņä£ļŖö ļæÉ ļ¬©ļŹĖņØś ĻĖ░ļ│ĖĻ░£ļģÉļ¦īņØä Ļ░äļץĒ׳ ņåīĻ░£ĒĢśĻ▓Āļŗż.
CNNņØĆ ņØ┤ļ»Ėņ¦Ćļź╝ ņØĖņŗØĒĢśņŚ¼ ļČäļźśĒĢśļŖö ļŹ░ ņŻ╝ļĪ£ ņé¼ņÜ®ļÉśļŖö ļ¬©ļŹĖļĪ£ ņØśļŻī ļČäņĢ╝ņØś ņśüņāü ļČäņäØņŚÉ ņØ┤ņÜ®ļÉśļŖö ļīĆĒæ£ņĀüņØĖ ļöźļ¤¼ļŗØ ĻĖ░ļ▓ĢņØ┤ļØ╝Ļ│Ā ĒĢĀ ņłś ņ׳ļŗż. ļöźļ¤¼ļŗØņØĆ ĻĘĖ ĒŖ╣ņä▒ņāü ļīĆņāü ņśüņāüņØś ĒŖ╣ņä▒Ļ│╝ ņāüĻ┤ĆņŚåņØ┤ ņØ╝ļ░śņĀüņØĖ ļ¬©ļŹĖļ¦üņØ┤ Ļ░ĆļŖźĒĢśļŗż. ĒŖ╣Ē׳ ņŗĀĻ▓Įļ¦ØņØś Ļ│äņĖĄņĀü ĻĄ¼ņĪ░ļŖö ņśüņāüņØś ņØ╝ļ░śņĀüņØĖ ĒŖ╣ņä▒ļōżļ┐Éļ¦ī ņĢäļŗłļØ╝ ņśüņāüņØś ņāēņØ┤ļéś ĒģīļæÉļ”¼ ņäĀĻ│╝ Ļ░ÖņØĆ ĒĢśņ£ä ņłśņżĆņØś ĒŖ╣ņä▒ļōżĻ╣īņ¦Ć ņŖżņŖżļĪ£ ņČöņČ£ĒĢśņŚ¼ ĒĢÖņŖĄĒĢĀ ņłś ņ׳ļŗż. CNNņŚÉņä£ļŖö ņØ┤ļ»Ėņ¦ĆņØś ņ£äņ╣ś, Ēü¼ĻĖ░, Ļ░üļÅä ļ│ĆĒÖö ļō▒ņŚÉ ņØśĒĢ┤ ņÖ£Ļ│ĪļÉśļŖö Ļ▓āĻ│╝ ņāüĻ┤ĆņŚåņØ┤ ņØ┤ļ»Ėņ¦Ćļź╝ ņØĖņŗØĒĢśĻĖ░ ņ£äĒĢ┤ ņ╗©ļ│╝ļŻ©ņģś(convolution)Ļ│╝ ĒÆĆļ¦ü(pooling) Ļ│╝ņĀĢņØä ļ░śļ│ĄņĀüņ£╝ļĪ£ ņłśĒ¢ēĒĢśļ®┤ņä£ ņØ┤ļ»Ėņ¦Ć ļŹ░ņØ┤Ēä░ņŚÉņä£ ņČöņāüĒÖöļÉ£ ņĀĢļ│┤ļź╝ ņČöņČ£ĒĢ£ļŗż. CNNņØĆ Ēü¼Ļ▓ī ļæÉ ļČĆļČäņ£╝ļĪ£ ĻĄ¼ņä▒ļÉśļŖöļŹ░, ņ┤łļ░śņŚÉļŖö ņ╗©ļ│╝ļŻ©ņģśĻ│╝ ĒÆĆļ¦ü Ļ│╝ņĀĢņØ┤ ļ░śļ│ĄņĀüņ£╝ļĪ£ ņĪ┤ņ×¼ĒĢśĻ│Ā, ļ¦łņ¦Ćļ¦ē ļČĆļČäņŚÉļŖö ļŗżņĖĄ ĒŹ╝ņģēĒŖĖļĪĀ ĻĄ¼ņĪ░ņØś fully connected layerļź╝ ļČÖņŚ¼ņä£ ļČäļźśĻĖ░(classifier)ņØś ĒśĢĒā£ļź╝ Ļ░¢Ļ▓ī ļÉ£ļŗż. ņ╗©ļ│╝ļŻ©ņģś ļŗ©Ļ│äņŚÉņä£ļŖö ņ×ģļĀźļÉ£ ņØ┤ļ»Ėņ¦ĆņŚÉ ņŚ¼ļ¤¼ Ļ░Ćņ¦Ć ĒĢäĒä░(filter)ļź╝ ņé¼ņÜ®ĒĢśņŚ¼ ņØ┤ļ»Ėņ¦ĆņØś ĒŖ╣ņ¦ĢņØä ļÅäņČ£ĒĢśĻ│Ā, ĒÆĆļ¦ü ļŗ©Ļ│äņŚÉņä£ļŖö ņØ┤ļ»Ėņ¦ĆņØś ĒŖ╣ņ¦ĢņØĆ ņ£Āņ¦ĆĒĢśļ®┤ņä£ ņØ┤ļ»Ėņ¦ĆņØś Ēü¼ĻĖ░ļź╝ ņżäņØ┤ļŖö ņŚŁĒĢĀņØä ĒĢ£ļŗż. ņØ┤ļ¤░ Ļ│╝ņĀĢņØä ņŚ¼ļ¤¼ ņ░©ļĪĆ ļ░śļ│ĄĒĢ┤ņä£ ĒĢśļéśņØś ņØ┤ļ»Ėņ¦ĆņŚÉņä£ ĻĘĖ ņØ┤ļ»Ėņ¦ĆņØś Ļ░£ļ│ä ĒŖ╣ņ¦ĢļōżņØä ļ│┤ņŚ¼ņŻ╝ļŖö ļŗżņ¢æĒĢ£ ņ×æņØĆ ņØ┤ļ»Ėņ¦ĆļōżņØä ļ¦īļōżņ¢┤ ļé┤Ļ▓ī ļÉ£ļŗż(Fig. 4). ĻĘĖļ”¼Ļ│Ā InceptionņØ┤ļéś ResNetĻ│╝ Ļ░ÖņØ┤ ĻĖ░ņĪ┤ņØś ņל ĒĢÖņŖĄļÉ£ CNN ļ¬©ļŹĖņØś ļé┤ļČĆņŚÉ ņ׳ļŖö ĒĢäĒä░ņģŗ(filter set)ļōżņØĆ ļŗżļźĖ ļ¼ĖņĀ£ ĒĢ┤Ļ▓░ņŚÉļÅä ņé¼ņÜ®ļÉĀ ņłś ņ׳ļŖöļŹ░ ņØ┤ļź╝ ņĀäņØ┤ ĒĢÖņŖĄ(transfer learning)ņØ┤ļØ╝Ļ│Ā ĒĢ£ļŗż. ņØ┤ļź╝ ņל ĒÖ£ņÜ®ĒĢśļ®┤ ņĀüņØĆ ņłśņØś ļŹ░ņØ┤Ēä░ļĪ£ļÅä ņÜ░ņłśĒĢ£ ņä▒ļŖźņØś CNN ļ¬©ļŹĖņØä ļ¦īļōż ņłś ņ׳ņ£╝ļ®░, ņĄ£ĻĘ╝ ņØśļŻī ļČäņĢ╝ņŚÉņä£ ļ░£Ēæ£ļÉśĻ│Ā ņ׳ļŖö ļöźļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņØ┤ļ»Ėņ¦Ć ļČäņäØ ņŚ░ĻĄ¼ļōżņØĆ ļīĆļČĆļČä ņØ┤ļ¤¼ĒĢ£ CNN ĻĖ░ļ▓ĢĻ│╝ ņĀäņØ┤ ĒĢÖņŖĄņØä ņØ┤ņÜ®ĒĢśĻ│Ā ņ׳ļŗż[8-10].
RNNņØĆ ņŗ£Ļ│äņŚ┤ ļŹ░ņØ┤Ēä░ņÖĆ Ļ░ÖņØ┤ ņŗ£Ļ░äņØś ĒØÉļ”äņŚÉ ļö░ļØ╝ ļ│ĆĒÖöĒĢśļŖö ļŹ░ņØ┤Ēä░ļź╝ ĒĢÖņŖĄĒĢśĻĖ░ ņ£äĒĢ£ ņØĖĻ│Ą ņŗĀĻ▓Įļ¦Øņ£╝ļĪ£ ļé┤ļČĆņŚÉ ņ×ÉĻĖ░ ņ×ÉņŗĀņ£╝ļĪ£ ļÉśļÅīņĢä ņśżļŖö ņĖĄ(layer)ņØä Ļ░Ćņ¦ĆĻ│Ā ņ׳ļŗż. ļö░ļØ╝ņä£ Ļ│╝Ļ▒░ņØś ņČ£ļĀź ļŹ░ņØ┤Ēä░ļź╝ ņ×¼ĻĘĆņĀüņ£╝ļĪ£ ņ░ĖņĪ░ĒĢ£ļŗż. ņŚ¼ĻĖ░ņŚÉņä£ ŌĆśņ×¼ĻĘĆŌĆÖļŖö Ēśäņ×¼ņØś Ļ▓░Ļ│╝Ļ░Ć ņØ┤ņĀä Ļ▓░Ļ│╝ņÖĆ ņŚ░Ļ┤Ćņä▒ņØä Ļ░Ćņ¦äļŗżļŖö ņØśļ»ĖņØ┤ļŗż. RNNņØĆ ņØīņä▒ņØĖņŗØ, ĻĖ░Ļ│äļ▓łņŚŁ, ņØ┤ļ»Ėņ¦Ć ņäżļ¬ģ ļō▒ ņŚ¼ļ¤¼ ļČäņĢ╝ņŚÉņä£ ĒÖ£ņÜ®ļÉśļŖö ļŹ░, ļīĆĒæ£ņĀüņØĖ ņśłĻ░Ć ļŗ©ņ¢┤ļéś ļ¼ĖņןņØä ņÖäņä▒ĒĢ┤ ņŻ╝ļŖö ņĢīĻ│Āļ”¼ņ”śņØ┤ļŗż. ņØ┤ņĀäņŚÉļŖö ņŚ¼ļ¤¼ ĻĖ░ņłĀņĀüņØĖ ļ¼ĖņĀ£ļĪ£ Ļ╣ŖņØĆ RNNņØĆ ĒĢÖņŖĄņØ┤ ļČłĻ░ĆļŖźĒ¢łņ£╝ļéś, ņĄ£ĻĘ╝ ņןļŗ©ĻĖ░ ĻĖ░ņ¢Ą(long short-term memory, LSTM) [11] ļ¬©ļŹĖĻ│╝ gated recurrent unit [11] ļ¬©ļŹĖņØ┤ ļ░£Ēæ£ļÉśļ®┤ņä£ Ļ░ĆļŖźĒĢśĻ▓ī ļÉśņŚłļŗż. ņĄ£ĻĘ╝ ņØśļŻī ļČäņĢ╝ņŚÉņä£ļÅä ņŗ£Ļ│äņŚ┤ņØä ņØ┤ņÜ®ĒĢśļŖö ļīĆļČĆļČäņØś ļöźļ¤¼ļŗØ ņŚ░ĻĄ¼ļōżņØĆ RNNņØś ĒĢ£ ņóģļźśņØĖ LSTM ļ¬©ļŹĖņØä ņØ┤ņÜ®ĒĢśĻ│Ā ņ׳ļŗż[12].
ļöźļ¤¼ļŗØņØś ĒĢ£Ļ│äļöźļ¤¼ļŗØ ļ¬©ļŹĖļōżņØ┤ ĻĖ░ņĪ┤ņØś ļ©ĖņŗĀļ¤¼ļŗØņØ┤ļéś ņØĖĻ│Ą ņŗĀĻ▓Įļ¦ØņØś ĒĢ£Ļ│äļź╝ ļø░ņ¢┤ ļäśņ£╝ļ®┤ņä£ ņØśļŻī ļČäņĢ╝ļź╝ ĒżĒĢ©ĒĢ£ ļ¦ÄņØĆ ļČäņĢ╝ņŚÉņä£ ĒÖ£ņÜ®ņØ┤ Ēü¼Ļ▓ī ņ”ØĻ░ĆĒĢśĻ│Ā ņ׳ņ¦Ćļ¦ī ņĢäņ¦ü ĒĢ┤Ļ▓░ņØ┤ ĒĢäņÜöĒĢ£ ņĀ£ĒĢ£ņĀÉļōżņØ┤ ņ׳ĻĖ░ļĢīļ¼ĖņŚÉ ĻĖ░ņĪ┤ņØś ļ©ĖņŗĀļ¤¼ļŗØ ļ¬©ļŹĖļōżņØä ņÖäņĀäĒ׳ ļīĆņ▓┤ĒĢśņ¦ĆļŖö ļ¬╗ĒĢśĻ│Ā ņ׳ļŗż. ļöźļ¤¼ļŗØņØ┤ Ļ░Ćņ¦ĆĻ│Ā ņ׳ļŖö ĒĢ£Ļ│äļŖö ņ▓½ņ¦Ė, ļöźļ¤¼ļŗØ ļ¬©ļŹĖņŚÉņä£ļŖö Ļ▓░Ļ│╝Ļ░Ć ņל ļéśņśżļŹöļØ╝ļÅä ļ¬©ļŹĖņØ┤ ņ¢┤ļ¢╗Ļ▓ī ĻĘĖ Ļ▓░Ļ│╝ļź╝ ļÅäņČ£ĒĢśņśĆļŖöņ¦ĆņŚÉ ļīĆĒĢ£ ĒĢ┤ņäØņØä ĒĢĀ ņłśļŖö ņŚåļŗż. ĻĘĖļלņä£ ņØ┤ļź╝ ļĖöļ×Öļ░ĢņŖż(black box) ļ¬©ļŹĖņØ┤ļØ╝Ļ│Ā ļČĆļź┤ĻĖ░ļÅä ĒĢ£ļŗż. ņśłļź╝ ļōżļ®┤, ĒÖśņ×ÉņØś ļŹ░ņØ┤Ēä░ļź╝ ĻĖ░ļ░śņ£╝ļĪ£ ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØ┤ ņØ┤ ĒÖśņ×ÉņŚÉĻ▓ī ĒĢäņÜöĒĢ£ ņ▓śļ░®ņØä ņśłņĖĪĒ¢łļŗżĒĢśļŹöļØ╝ļÅä, ņÖ£ ĻĘĖļ¤░ ņ▓śļ░®ņØ┤ ļéśņÖöļŖöņ¦Ć ņäżļ¬ģĒĢśņ¦Ć ļ¬╗ĒĢśĻĖ░ ļĢīļ¼ĖņŚÉ ņØśņé¼ņÖĆ ĒÖśņ×É ļ¬©ļæÉ ĻĘĖ Ļ▓░Ļ│╝ļź╝ ļ░øņĢäļōżņØ┤ĻĖ░ Ēלļōż ņłś ņ׳ļŗż. ļæśņ¦Ė, ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØä ĒĢÖņŖĄņŗ£ĒéżĻĖ░ ņ£äĒĢ┤ņä£ļŖö ņØ╝ļ░śņĀüņ£╝ļĪ£ ļ¦ÄņØĆ ņ¢æņØś ļŹ░ņØ┤Ēä░Ļ░Ć ĒĢäņÜöĒĢśĻĖ░ ļĢīļ¼ĖņŚÉ, ļ¼ĖņĀ£ņŚÉ ļö░ļØ╝ ļŗżļź┤ņ¦Ćļ¦ī ļŹ░ņØ┤Ēä░Ļ░Ć ņĀüņØĆ Ļ▓ĮņÜ░ņŚÉļŖö ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØä ņĀüņÜ®ĒĢśĻĖ░ ņ¢┤ļĀżņÜ┤ Ļ▓ĮņÜ░Ļ░Ć ļ¦Äļŗż. ĒŖ╣Ē׳ ņ×äņāüņŚÉņä£ļŖö ņŚ¼ļ¤¼ ņĀ£ĒĢ£ļōż ļĢīļ¼ĖņŚÉ ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØä ļ¦īļōżĻĖ░ņŚÉ ĒĢäņÜöĒĢ£ ļŹ░ņØ┤Ēä░ņØś ņ¢æņØä ņČ®ņĪ▒ņŗ£Ēéżņ¦Ć ļ¬╗ĒĢĀ ļĢīĻ░Ć ļ¦Äļŗż. ņģŗņ¦Ė, ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØä ĒĢÖņŖĄņŗ£ĒéżĻĖ░ ņ£äĒĢ┤ņä£ļŖö ņŚäņ▓Łļé£ Ļ│äņé░ļ¤ēņØ┤ ĒĢäņÜöĒĢśļ®░, ņØ┤ļź╝ Ļ░Éļŗ╣ĒĢśĻĖ░ ņ£äĒĢ┤ņä£ Ļ│Āņé¼ņ¢æņØś ņ╗┤Ēō©Ēä░ ĒĢśļō£ņø©ņ¢┤ņÖĆ ņāüļīĆņĀüņ£╝ļĪ£ ĻĖ┤ ĒĢÖņŖĄ ņŗ£Ļ░äņØä ņÜöĻĄ¼ĒĢ£ļŗż. ļö░ļØ╝ņä£, Ļ▓░Ļ│╝ņØś ĒĢ┤ņäØņØ┤ ĒĢäņÜöĒĢśĻ▒░ļéś, ļŹ░ņØ┤Ēä░ņØś ņ¢æņØ┤ ņĀüņØĆ Ļ▓ĮņÜ░, ĻĘĖļ”¼Ļ│Ā ņ¦¦ņØĆ ĒĢÖņŖĄ ņŗ£Ļ░äņØ┤ ĒĢäņÜöĒĢĀ ļĢīļŖö ĻĖ░ņĪ┤ņØś ļ©ĖņŗĀļ¤¼ļŗØ ļ¬©ļŹĖņØ┤ ļöźļ¤¼ļŗØ ļ¬©ļŹĖļ│┤ļŗż ĒÜ©Ļ│╝ņĀüņØ╝ ņłś ņ׳ļŗż.
ņ×äņāüņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØś ņĀüņÜ®ļ©ĖņŗĀļ¤¼ļŗØņØ┤ļéś ļöźļ¤¼ļŗØĻ│╝ Ļ░ÖņØĆ ņØĖĻ│Ąņ¦ĆļŖź ĻĖ░ņłĀņØĆ ņØśļŻī ļČäņĢ╝ņØś ņŚ¼ļ¤¼ ļČäņĢ╝ņŚÉņä£ ĒÖ£ņÜ®ļÉĀ ņłś ņ׳ļŗż. ĒŖ╣Ē׳ ņĄ£ĻĘ╝ ļöźļ¤¼ļŗØ ĻĖ░ļ▓ĢņØś ļ░£ļŗ¼ļĪ£ ņØ┤ļ»Ėņ¦Ć ļČäņäØņØś ņä▒ļŖźņØ┤ ļåÆņĢäņ¦Ćļ®┤ņä£ ņśüņāü ļČäņĢ╝ņŚÉņä£ ļ¦ÄņØ┤ ņĀüņÜ®ļÉśņ¢┤ ļ¦ÄņØĆ ņŚ░ĻĄ¼Ļ░Ć ņ¦äĒ¢ē ņżæņØ┤ļ®░ ņØ╝ļČĆļŖö ņŗżņĀ£ ņ×äņāüņŚÉ ņé¼ņÜ®ļÉśĻ│Ā ņ׳ļŗż. ņśüņāü ļČäņĢ╝ņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØś ņŚŁĒĢĀņØĆ Ēü¼Ļ▓ī ņØ┤ļ»Ėņ¦Ćļź╝ ĒåĄĒĢ┤ ĒŖ╣ņĀĢ ņ¦łĒÖś ņŚ¼ļČĆļź╝ ĻĄ¼ļČäĒĢĀ ņłś ņ׳ļŖö ļČäļźś, ņØ┤ļ»Ėņ¦ĆņŚÉņä£ ĒŖ╣ņĀĢ ņןĻĖ░ļéś ļ│æļ│ĆņØä ņ×ÉļÅÖņ£╝ļĪ£ ļČäļ”¼ĒĢĀ ņłś ņ׳ļŖö ļČäĒĢĀ(segmentation)Ļ│╝ ņ╗┤Ēō©Ēä░ļ│┤ņĪ░Ļ▓ĆņČ£(computer-assisted detection) ļ░®ņŗØņØś ļ│æļ│Ć ņ×ÉļÅÖ Ļ▓ĆņČ£, ĻĘĖļ”¼Ļ│Ā ņĀĆĒÆłņ¦łņØś ņśüņāüņŚÉņä£ Ļ│ĀĒÆłņ¦łņØś ņśüņāüĻ│╝ ņ£Āņé¼ĒĢ£ ņśüņāüņØä ļ¦īļōżņ¢┤ļé┤ļŖö ļ¬©ņØś(simulation) ļō▒ņØ┤ ņ׳ļŗż[13]. Ļ░£ņØĖļ│ä ļ¦×ņČż ņ╣śļŻīļź╝ ņČöĻĄ¼ĒĢśļŖö ņĀĢļ░ĆņØśļŻī(precision medicine)Ļ░Ć ņØśļŻī ļČäņĢ╝ņØś ĒÖöļæÉĻ░Ć ļÉśĻ│Ā ņ׳ļŖö Ēśä ņāüĒÖ®ņŚÉņä£, ļ╣ģļŹ░ņØ┤Ēä░ņØś ļČäņäØņØä ĒåĄĒĢ┤ ņ╣śļŻīņØś Ļ▓░Ļ│╝ ļō▒ņØä ļ»Ėļ”¼ ņśłņĖĪĒĢĀ ņłś ņ׳ļŖö ļČäļźś ļČäņĢ╝ņŚÉņä£ Ļ░Ćņן ĒÖ£ļ░£ĒĢśĻ▓ī ļ©ĖņŗĀļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼Ļ░Ć ņ¦äĒ¢ēņżæņØ┤ļŗż.
ņØ┤ļ╣äņØĖĒøäĻ│╝ ņśüņŚŁņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ņØ┤ļ╣äņØĖĒøäĻ│╝ ļČäņĢ╝ļÅä ļŗżļźĖ ņØśļŻī ļČäņĢ╝ņÖĆ ļ¦łņ░¼Ļ░Ćņ¦ĆļĪ£ ļ©ĖņŗĀļ¤¼ļŗØņØ┤ļéś ļöźļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļōżņØ┤ ņ¦äĒ¢ēņżæņØ┤ļŗż. ĻĘĖ ņżæņŚÉņä£ļÅä ļæÉĻ▓ĮļČĆ ņśüņŚŁņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼Ļ░Ć Ļ░Ćņן ĒÖ£ļ░£ĒĢ£ļŹ░, CNN ļ¬©ļŹĖņØä ņØ┤ņÜ®ĒĢśņŚ¼ ļé┤ņŗ£Ļ▓ĮņØ┤ļéś CT, MRI Ļ░ÖņØĆ ņśüņāüņŚÉņä£ ņĢö ļ│æļ│Ć ļČĆņ£äļź╝ ņ×ÉļÅÖņ£╝ļĪ£ Ļ░Éņ¦ĆĒĢśņŚ¼ ņĀäņ▓┤ ņóģņ¢æņØś ļČĆĒö╝ļź╝ Ļ│äņé░ĒĢśĻ▒░ļéś[14,15], ĒøäļæÉļé┤ņŗ£Ļ▓ĮņŚÉņä£ ļ│æļ│ĆņŚÉ ļö░ļØ╝ ņ×ÉļÅÖņ£╝ļĪ£ ņ¦łĒÖśņØä ļČäļźśĒĢśĻĖ░ļÅä ĒĢ£ļŗż[16-19]. ņØ┤ņÖĆ Ļ░ÖņØ┤ ņØ┤ļ»Ėņ¦Ćļéś ņśüņāüļ¦īņØä Ļ░Ćņ¦ĆĻ│Ā ņ¦łĒÖśņØä ņ×ÉļÅÖ ņ¦äļŗ©(automated detection)ĒĢśĻĖ░ ņ£äĒĢ£ ņŚ░ĻĄ¼ļōżņØ┤ Ļ┤æĒĢÖņāØĻ▓Ć(optical biopsy)ņØ┤ļØ╝ļŖö ņØ┤ļ”äņ£╝ļĪ£ ņŗ£Ē¢ēņżæņØ┤ļŗż[20]. ņØ┤ļ░¢ņŚÉ ļ©ĖņŗĀļ¤¼ļŗØ ņĢīĻ│Āļ”¼ņ”śņØä ņØ┤ņÜ®ĒĢśņŚ¼ ļæÉĻ▓ĮļČĆņĢö ĒÖśņ×ÉņØś ņāØņĪ┤ņ£©ņØ┤ļéś ĒĢ®ļ│æņ”Ø ļ░£ņāØ, Ļ▓ĮļČĆ ļ”╝ĒöäņäĀ ņĀäņØ┤, ņ×¼ļ░£ ņŚ¼ļČĆ ļō▒ņØä ņśłņĖĪĒĢśļŖö ņŚ░ĻĄ¼ļōżļÅä ņŗ£Ē¢ēļÉśĻ│Ā ņ׳ļŗż[21-28]. ņØ┤Ļ│╝ ņśüņŚŁņŚÉņä£ļŖö ļ©ĖņŗĀļ¤¼ļŗØ ĻĖ░ļ▓ĢņØä ņØ┤ņÜ®ĒĢśņŚ¼ ļÅīļ░£ņä▒ ļé£ņ▓Ł ĒÖśņ×ÉņØś ņśłĒøäļéś[29,30], ņ▓ŁņŗĀĻ▓Įņ┤łņóģ ņłśņłĀ ĒÖśņ×ÉņŚÉņä£ ņ▓ŁļĀź ļ│┤ņĪ┤ņØ┤ļéś ņ×¼ļ░£ ņŚ¼ļČĆ[31,32], ĻĘĖļ”¼Ļ│Ā ņØĖĻ│ĄņÖĆņÜ░ ņłśņłĀ Ļ▓░Ļ│╝ļź╝ ņśłņĖĪĒĢśĻĖ░ļÅä ĒĢ£ļŗż[33]. ĻĘĖļ”¼Ļ│Ā CNN ļ¬©ļŹĖņØä ĒåĄĒĢ┤ņä£ ņØ┤ļé┤ņŗ£Ļ▓ĮņØ┤ļéś ņĢłļ®┤ ņé¼ņ¦ä, CT/MRI ņØ┤ļ»Ėņ¦Ćļź╝ ļČäņäØĒĢśņŚ¼ Ļ│Āļ¦ē ņ▓£Ļ│ĄņØ┤ļéś ņżæņØ┤ņŚ╝, ņÖĖņØ┤ĻĖ░ĒśĢ, ņŗĀĻ▓Įņ┤łņóģ ļō▒ņØä ņ×ÉļÅÖ ņ¦äļŗ©ĒĢśļŖö ņŚ░ĻĄ¼ļōżļÅä ņŗ£Ē¢ēļÉśņŚłļŗż[34-39].
ļ╣äĻ│╝ ņśüņŚŁņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØ ņŚ░ĻĄ¼ļ╣äĻ│╝ ņśüņŚŁņŚÉņä£ļÅä ļ©ĖņŗĀļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļōżņØ┤ ĒÖ£ļ░£Ē׳ ņ¦äĒ¢ē ņżæņØ┤ļŗż. ņ┤łĻĖ░ņŚÉļŖö ļ©ĖņŗĀļ¤¼ļŗØ ĻĖ░ļ▓ĢņØä ņØ┤ņÜ®ĒĢśņŚ¼ ļ¦īņä▒ ļČĆļ╣äļÅÖņŚ╝ņØś Ēæ£ĒśäĒśĢ(phenotype)ņØä ļČäļźśĒĢśļŖö ņŚ░ĻĄ¼Ļ░Ć ņŻ╝ļÉ£ ĒØÉļ”äņØ┤ņŚłņ£╝ļéś, ĻĘ╝ļלņŚÉļŖö CNNĻ│╝ Ļ░ÖņØĆ ļöźļ¤¼ļŗØ ĻĖ░ļ▓ĢņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļōżņØ┤ ņĀÉņ░© ļŖśĻ│Ā ņ׳ļŖö ņČöņäĖņØ┤ļŗż(Table 2).
ļ¦īņä▒ļČĆļ╣äļÅÖņŚ╝ ĒÖśņ×ÉļōżņŚÉĻ▓ī ļ¦×ņČżĒśĢ ņ╣śļŻī ņĀäļץņØä ĻĄ¼ņé¼ĒĢśĻĖ░ ņ£äĒĢ┤ņä£ļŖö ĒÖśņ×ÉļōżņØś ņ×äņāü ņåīĻ▓¼ņŚÉ ļö░ļźĖ Ēæ£ĒśäĒśĢļ┐Éļ¦ī ņĢäļŗłļØ╝, ļČäņ×ÉņłśņżĆņØś ļ®┤ņŚŁĒĢÖņĀü ĒŖ╣ņä▒ņØä ļ░śņśüĒĢ£ ļé┤ņ×¼ĒśĢ(endotype)Ļ╣īņ¦Ć Ļ│ĀļĀżļÉśņ¢┤ņĢ╝ ĒĢ£ļŗż. ņØ┤ļź╝ ņ£äĒĢ┤ņä£ ļŗżņ¢æĒĢ£ ļ░öņØ┤ņśżļ¦łņ╗ż(biomarker)ļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ļ¦īņä▒ļČĆļ╣äļÅÖņŚ╝ņØä ļČäļźśĒĢśļŖö ņŗ£ļÅäļōżņØ┤ ņ׳ņ¢┤ ņÖöļŗż. ņØ┤ļź╝ ņ£äĒĢ┤ ļ©ĖņŗĀļ¤¼ļŗØņ£╝ļĪ£ ļ╣ģļŹ░ņØ┤Ēä░ļź╝ ļŗżļŻ©ļŖö ņĀæĻĘ╝ ļ░®ļ▓ĢņØä ĒåĄĒĢ┤ ļ¦īņä▒ļČĆļ╣äļÅÖņŚ╝ņØä ļČäļźśĒĢśĻ│Āņ×É ĒĢśļŖö ņŗ£ļÅäļōżņØ┤ ņ׳ņ¢┤ ņÖöļŖöļŹ░, ļīĆļČĆļČäņØś ņŚ░ĻĄ¼Ļ░Ć ĻĄ░ņ¦æĒÖö ņĢīĻ│Āļ”¼ņ”ś(clustering algorhithm)ņØä ņØ┤ņÜ®ĒĢśņśĆļŗż. ĻĄ░ņ¦æĒÖö ņĢīĻ│Āļ”¼ņ”śņØĆ ļŹ░ņØ┤Ēä░ļź╝ ĒŖ╣ņä▒ņØś ņ£Āņé¼ĒĢ©ņŚÉ ļö░ļØ╝ ņØ╝ņĀĢ ņłśņØś ĻĄ░ņ¦æņ£╝ļĪ£ ļČäļźśĒĢśļŖö ņĢīĻ│Āļ”¼ņ”śņ£╝ļĪ£, ņØ┤ļĢī ņ×ģļĀźļÉ£ ļŹ░ņØ┤Ēä░ļŖö ĒŖ╣ņä▒Ļ░Æļ¦ī ņĪ┤ņ×¼ĒĢśĻ│Ā Ļ▓░Ļ│╝Ļ░ÆņØ┤ ņĪ┤ņ×¼ĒĢśņ¦Ć ņĢŖņ£╝ļ»ĆļĪ£ ļ©ĖņŗĀļ¤¼ļŗØņØś ļ╣äņ¦ĆļÅä ĒĢÖņŖĄ(unsupervised learning)ņŚÉ ĒĢ┤ļŗ╣ĒĢ£ļŗż. ļ©ĖņŗĀļ¤¼ļŗØņØ┤ ņĀüņÜ®ļÉ£ ļ¦īņä▒ļČĆļ╣äļÅÖņŚ╝ ļČäļźś ņŚ░ĻĄ¼ņŚÉņä£ļŖö ļŹ░ņØ┤Ēä░ņØś ĒŖ╣ņä▒ņŚÉ ļö░ļØ╝ ņ”Øņāü, ļ╣äņÜ®ņóģ, ĒØĪņŚ░ļĀź, ņ▓£ņŗØ ņ£Āļ¼┤ ļō▒Ļ│╝ Ļ░ÖņØĆ ņ×äņāü ņåīĻ▓¼ņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ņÖĆ ņäĖĒżĒæ£ņ¦Ćņ×É(cell marker)ļéś ņé¼ņØ┤ĒåĀņ╣┤ņØĖ(cytokine)Ļ│╝ Ļ░ÖņØĆ ļ░öņØ┤ņśżļ¦łņ╗żļź╝ ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļĪ£ ļéśļłī ņłś ņ׳ļŗż. ņ×äņāü ņåīĻ▓¼ņØä ļŹ░ņØ┤Ēä░ ĒŖ╣ņä▒ņ£╝ļĪ£ ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļź╝ ņé┤ĒÄ┤ļ│┤ļ®┤, Soler ļō▒[40]ņØĆ ņĢĮļ¼╝ņŚÉ ļ░śņØæĒĢśņ¦Ć ņĢŖļŖö 382ļ¬ģņØś ļ¦īņä▒ļČĆļ╣äļÅÖņŚ╝ ĒÖśņ×ÉņŚÉņä£ 32Ļ░£ņØś ĒŖ╣ņä▒ņØä ņé¼ņÜ®ĒĢśņŚ¼ ĻĄ░ņ¦æĒÖö ļČäņäØņØä ņŗ£Ē¢ēĒ¢łņØä ļĢī, ļīĆņāü ĒÖśņ×ÉļōżņØ┤ 5Ļ░£ņØś ĻĄ░ņ¦æņ£╝ļĪ£ ļČäļźśļÉśņŚłĻ│Ā, ļéśņØ┤, ņāØņé░ņä▒ ņĀĆĒĢś, Sino-nasal Outcome Test(SNOT-22) ņĀÉņłśĻ░Ć ĻĄ░ņ¦æņØä ļČäļźśĒĢśļŖö ļŹ░ Ļ░Ćņן ņØśļ»Ėņ׳ļŖö ĒŖ╣ņä▒ņØ┤ņŚłļŗż. ĒĢśņ¦Ćļ¦ī ļ╣äņÜ®ņóģ ņ£Āļ¼┤, ņĢäĒåĀĒö╝, ņ▓£ņŗØ, ņĢäņŖżĒö╝ļ”░ Ļ│╝ļ»╝ņä▒ ļō▒Ļ│╝ Ļ░ÖņØ┤ ĻĖ░ņĪ┤ņŚÉ ļ¦īņä▒ ļČĆļ╣äļÅÖņŚ╝ņØś Ēæ£ĒśäĒśĢņØä ĻĄ¼ļČäĒĢśļŖö ļŹ░ ņżæņÜöĒĢśĻ▓ī ņŚ¼Ļ▓©ņĪīļŹś ĒŖ╣ņä▒ļōżņØĆ ĻĘĖ ņżæņÜöļÅäĻ░Ć ļé«ņØĆ Ļ▓āņ£╝ļĪ£ ļ│┤Ļ│ĀĒĢśņśĆļŗż. Lal ļō▒[41]ņØĆ ļ╣äņÜ®ņóģņØä ļÅÖļ░śĒĢśņ¦Ć ņĢŖņØĆ 146ļ¬ģņØś ĒÖśņ×ÉņŚÉņä£ SNOT-22 ņĀÉņłśņŚÉ ļö░ļØ╝ ĻĄ░ņ¦æ ļČäņäØņØä ņŗ£Ē¢ēĒĢśņśĆļŖöļŹ░, 4Ļ░£ņØś ĻĄ░ņ¦æ(severe, moderate with sinonasal symptoms, moderate with pychological sleep symptoms, mild)ņ£╝ļĪ£ ļéśļłī ņłś ņ׳ņ£╝ļ®░, severe ĻĄ░ņ¦æņØś Ļ▓ĮņÜ░ņŚÉļŖö, ļ╣äņÜ®ņóģ ĒÖśņ×ÉļōżĻ│╝ ļ¦łņ░¼Ļ░Ćņ¦ĆļĪ£ ņ▓£ņŗØ ļ░Å ņĪ░ņ¦üļé┤ ĒśĖņé░ĻĄ¼(tissue eosinophilia)ņÖĆ Ļ░ĢĒĢ£ ņāüĻ┤Ć Ļ┤ĆĻ│äļź╝ ļ│┤ņśĆļŗż. ļ░öņØ┤ņśżļ¦łņ╗żļōżņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļōżņØä ļ│┤ļ®┤, Tomassen ļō▒[42]ņØ┤ ņłśņłĀ Ēøä ņĪ░ņ¦üņŚÉņä£ ņé¼ņØ┤ĒåĀņ╣┤ņØĖņØä ņØ┤ņÜ®ĒĢ£ ĻĄ░ņ¦æ ļČäņäØņØä ņŗ£Ē¢ēĒĢśņśĆļŗż. ļīĆņāü ĒÖśņ×ÉļōżņØĆ 10Ļ░£ņØś ĻĄ░ņ¦æņ£╝ļĪ£ ļČäļźśļÉśņŚłĻ│Ā, ņØ┤ ņżæ 3Ļ░£ņØś ĻĄ░ņ¦æņØ┤ ļ╣äņÜ®ņóģĻ│╝ ņ▓£ņŗØņŚÉ Ļ░ĢĒĢ£ ņāüĻ┤ĆĻ┤ĆĻ│äļź╝ ļ│┤ņśĆņ¦Ćļ¦ī ļéśļ©Ėņ¦Ć 3Ļ░£ ĻĄ░ņ¦æņŚÉņä£ļŖö ļ░śļīĆņØś Ļ▓░Ļ│╝ļź╝ ļ│┤ņśĆļŗż. Kim ļō▒[43]ņØĆ ļ╣äņÜ®ņóģņØä Ļ░Ćņ¦ĆĻ│Ā ņ׳ļŖö 375ļ¬ģņØś ĒÖśņ×ÉņŚÉņä£ ņĀÉļ¦ēĻ│╝ ĒśłņżæņØś ĒśĖņé░ĻĄ¼ ņłś, ļéśņØ┤, CTņåīĻ▓¼, ņ▓£ņŗĀ ņ£Āļ¼┤ ļō▒ 6Ļ░£ņØś ĒŖ╣ņä▒ņØä Ļ░Ćņ¦ĆĻ│Ā ĻĄ░ņ¦æ ļČäņäØņØä ņŗ£Ē¢ēĒĢśņŚ¼, 6Ļ░£ņØś ĻĄ░ņ¦æņ£╝ļĪ£ ļČäļźśĒĢśņśĆļŗż. ņØ┤ ņżæ 2Ļ░£ņØś ĻĄ░ņ¦æņŚÉņä£ļŖö ņ▓£ņŗØĻ│╝ Ļ┤ĆļĀ©ņØ┤ ļÉśņŚłņ¦Ćļ¦ī, ļéśļ©Ėņ¦Ć 4Ļ░£ņØś ĻĄ░ņ¦æņŚÉņä£ļŖö ņāüĻ┤Ćņä▒ņØ┤ ņŚåņŚłļŗż. ņØ┤ņāüņØś ĻĄ░ņ¦æ ļČäņäØ ņŚ░ĻĄ¼ļōżņŚÉņä£ ņ×äņāü ņåīĻ▓¼ ĒŖ╣ņä▒ņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ņÖĆ ļ░öņØ┤ņśżļ¦łņ╗żļź╝ ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļōżņØś Ļ▓░Ļ│╝ļź╝ ļ╣äĻĄÉĒĢ┤ ļ│┤ļ®┤, ļ░öņØ┤ņśżļ¦łņ╗żļź╝ ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ļōżņŚÉņä£ļŖö ļ╣äņÜ®ņóģĻ│╝ ņŗ¼ĒĢ£ ņ”Øņāü ļ░Å ņŚ╝ņ”Ø ņé¼ņØ┤ņŚÉ Ļ░ĢĒĢ£ ņāüĻ┤ĆĻ┤ĆĻ│äļź╝ ļ│┤ņØĖ ļ░śļ®┤, ņ×äņāü ņåīĻ▓¼ņØä ņØ┤ņÜ®ĒĢ£ ņŚ░ĻĄ¼ņŚÉņä£ļŖö ļ░śļīĆļĪ£ ļæś Ļ░äņŚÉ ņāüĻ┤ĆĻ┤ĆĻ│äĻ░Ć ļé«ņØĆ Ļ▓āņ£╝ļĪ£ ļ│┤ņŚ¼ņ¦äļŗż.
ņØ┤ļ¤¼ĒĢ£ ļ©ĖņŗĀļ¤¼ļŗØ Ļ┤ĆļĀ© ņŚ░ĻĄ¼ļōżņØĆ ļ¦īņä▒ļČĆļ╣äļÅÖņŚ╝ņØś ļČäļźśņŚÉņä£ ĻĖ░ņĪ┤ ņŚ░ĻĄ¼ļōżĻ│╝ļŖö ņØ╝ļČĆ ņāüļ░śļÉśļŖö Ļ▓░Ļ│╝ļōżņØä ļ│┤ņŚ¼ ņŻ╝Ļ│Ā ņ׳ļŗż. ĒĢśņ¦Ćļ¦ī, ļ©ĖņŗĀļ¤¼ļŗØ ļ¬©ļŹĖ ņ×Éņ▓┤ņØś ĒŖ╣ņä▒ņāü ļ¬ć Ļ░Ćņ¦Ć ņĀ£ĒĢ£ņĀÉļōżņØ┤ ņ׳ņ£╝ļ»ĆļĪ£ ĒĢ┤ņäØņŚÉ ņ£ĀņØśĒĢ┤ņĢ╝ ĒĢĀ ĒĢäņÜöĻ░Ć ņ׳ļŗż. ņÜ░ņäĀ, ņŚ░ĻĄ¼ ļīĆņāü ĒÖśņ×ÉļōżņØś ņłśĻ░Ć ņØ╝ļ░śņĀüņØĖ ļ╣ģļŹ░ņØ┤Ēä░ņŚÉ ļ╣äĒĢ┤ ņāüļīĆņĀüņ£╝ļĪ£ ņĀüļŗżļŖö ņĀÉņØ┤ļŗż. ņØ╝ļ░śņĀüņ£╝ļĪ£ ņØ┤ļ╣äņØĖĒøäĻ│╝, ĒŖ╣Ē׳ ļ╣äĻ│╝ ņśüņŚŁņØś ņ×äņāü ņŚ░ĻĄ¼ņŚÉņä£ ļ╣ģļŹ░ņØ┤Ēä░ļĪ£ ļČĆļź╝ ņłś ņ׳ņØä ļ¦īĒü╝ ņČ®ļČäĒĢ£ ļŹ░ņØ┤Ēä░ļź╝ ļ¬©ņ£╝ļŖö Ļ▓āņØĆ ĒśäņŗżņĀüņ£╝ļĪ£ ņ¢┤ļĀżņÜ┤ Ļ▓ĮņÜ░Ļ░Ć ļ¦ÄĻĖ░ ļĢīļ¼ĖņŚÉ, ņØ┤ļōż ņŚ░ĻĄ¼ļōżņØś Ļ▓░Ļ│╝ļź╝ ĒĢ┤ņäØĒĢ©ņŚÉ ņ׳ņ¢┤ ņØ┤ņóģ ņśżļźś(type II error)ņØś Ļ░ĆļŖźņä▒ņØä ĒĢŁņāü ņŚ╝ļæÉņŚÉ ļæÉņ¢┤ņĢ╝ ĒĢ£ļŗż. ļśÉĒĢ£, ņŚ░ĻĄ¼ņ×ÉĻ░Ć ļīĆņāü ĒÖśņ×ÉņÖĆ ļŹ░ņØ┤Ēä░ņØś ĒŖ╣ņä▒ņØä ņ¢┤ļ¢╗Ļ▓ī ņäĀĒāØĒĢśļŖöĻ░ĆņŚÉ ļö░ļØ╝ ļ¬©ļŹĖņØś ņä▒ļŖź ļ┐É ņĢäļŗłļØ╝ Ļ▓░Ļ│╝ņŚÉļÅä ņśüĒ¢źņØä ļü╝ņ╣Ā ņłś ņ׳ņØīņØä ņØ┤ĒĢ┤ĒĢ┤ņĢ╝ ĒĢ£ļŗż[44].
ļ╣äĻ│╝ņśüņŚŁņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØä ņØ┤ņÜ®ĒĢ£ ļŗżļźĖ ļČäņĢ╝ļŖö ņŚ¼ļ¤¼ ļöźļ¤¼ļŗØ ĻĖ░ļ▓ĢņØä ņØ┤ņÜ®ĒĢśņŚ¼ ņśüņāü ņØ┤ļ»Ėņ¦Ćļź╝ ņ×ÉļÅÖ ļČäļźśĒĢ┤ ļé┤ļŖö ņŚ░ĻĄ¼ļōżņØ┤ļŗż. ņĄ£ĻĘ╝ Chowdhury ļō▒[45]ņØĆ CNN ņĢīĻ│Āļ”¼ņ”śņØä ņØ┤ņÜ®ĒĢśņŚ¼ ļČĆļ╣äļÅÖ CTņŚÉņä£ osteomeatal complexņØś ĒÅÉņćä ņŚ¼ļČĆļź╝ ņ×ÉļÅÖņ£╝ļĪ£ ļČäļźśĒĢ┤ ļé┤ļŖö ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØä ļ│┤Ļ│ĀĒĢśņśĆļŗż. ņØ┤ ņŚ░ĻĄ¼ņŚÉņä£ļŖö ļ¦īņä▒ļČĆļ╣äļÅÖņŚ╝ ĒÖśņ×É 239ļ¬ģņØś ļČĆļ╣äļÅÖ CTņŚÉņä£ ņČöņČ£ĒĢ£ 956Ļ░£ņØś Ļ┤Ćņāü(coronal) ņØ┤ļ»Ėņ¦ĆņŚÉ osteomeatal complexņØś ĒÅÉņćä ņŚ¼ļČĆļź╝ labellingĒĢ£ ļŗżņØī, ĻĖ░ņĪ┤ņŚÉ ņØ┤ļ»Ė 128ļ¦ī ņןņØ┤ ļäśļŖö ņØ┤ļ»Ėņ¦ĆļōżļĪ£ ĒĢÖņŖĄļÉśņ¢┤ ņ׳ļŖö GoogleņØś Inception-V3 CNN ļ¬©ļŹĖņØä ĒåĄĒĢ┤ ņĀäņØ┤ ĒĢÖņŖĄņØä ņŗ£Ē¢ēĒĢśņśĆļŗż. ĻĘĖ Ļ▓░Ļ│╝, osteomeatal complexņØś ĒÅÉņćäņŚ¼ļČĆ ņ¦äļŗ©ņŚÉņä£ 85%[95% confidence interval(CI), 78~92%]ņØś ņĀĢĒÖĢļÅäļź╝ ļ│┤ņśĆĻ│Ā area under the receiver operating characteristics curve (AUC)Ļ░ÆņØĆ 0.87ņØ┤ņŚłļŗż. Huang ļō▒[46]ņØĆ 675Ļ░£ņØś ļČĆļ╣äļÅÖ CT ņØ┤ļ»Ėņ¦Ćļź╝ Inception V3 ļ¬©ļŹĖļĪ£ ņĀäņØ┤ ĒĢÖņŖĄņŗ£Ēé© Ļ▓░Ļ│╝, CTņāüņŚÉņä£ ņĀäņé¼Ļ│©ļÅÖļ¦ź(anterior ethmoidal artery)ņØś ņ£äņ╣śļź╝ 82.7%ņØś ņĀĢĒÖĢļÅäņÖĆ AUC 0.86ņ£╝ļĪ£ ļČäļźśĒĢ┤ ļé╝ ņłś ņ׳ņŚłļŗż. ņØ┤ļ░¢ņŚÉ ņĄ£ĻĘ╝ņŚÉļŖö 690ļ¬ģņØś ĒÖśņ×ÉņŚÉņä£ ņŗ£Ē¢ēļÉ£ ļČĆļ╣äļÅÖ CTļź╝ CNN ļ¬©ļŹĖļĪ£ ĒĢÖņŖĄņŗ£ņ╝£ņä£, CTņāüņŚÉņä£ ļČĆļ╣äļÅÖ ļČĆļČäļ¦īņØä ļČäĒĢĀĒĢśņŚ¼ ļČĆļ╣äļÅÖņØś ņĀäņ▓┤ ļČĆĒö╝ņÖĆ Ēś╝Ēāü ņĀĢļÅäļź╝ ņ×ÉļÅÖņ£╝ļĪ£ Ļ│äņé░ĒĢśļŖö ļ¬©ļŹĖņØ┤ ļ│┤Ļ│ĀļÉśĻĖ░ļÅä ĒĢśņśĆļŗż[47].
ļöźļ¤¼ļŗØ ĻĖ░ļ▓ĢņØä ņØ┤ņÜ®ĒĢśņŚ¼ ļ╣äņØĖļæÉņĢöļź╝ ņ¦äļŗ©ĒĢśĻ│Āņ×É ĒĢśļŖö ņŚ░ĻĄ¼ļōżļÅä ņŗ£Ē¢ēļÉśņŚłļŗż. Li ļō▒[48]ņØĆ 27536Ļ░£ņØś ļ╣äņØĖļæÉ ļé┤ņŗ£Ļ▓Į ņØ┤ļ»Ėņ¦Ćļź╝ CNN ļ¬©ļŹĖņØä ĒåĄĒĢ┤ ĒĢÖņŖĄņŗ£ņ╝░Ļ│Ā, ļ╣äņØĖļæÉņĢö ņ¦äļŗ©ņŚÉņä£ 88.0%(95% CI, 86.1~89.6%)ņØś ņĀĢĒÖĢļÅäļź╝ ļ│┤ņŚ¼ ņĀäļ¼ĖņØś(80.5%)ļéś ņĀäĻ│ĄņØś(72.8%)ļ│┤ļŗż ņÜ░ņłśĒĢ£ Ļ▓░Ļ│╝ļź╝ ļéśĒāĆļé┤ņŚłļŗż. ļśÉĒĢ£ ņØ┤ ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØ┤ ļ╣äņØĖļæÉņĢöņØś ļ│æļ│Ć ļČĆņ£äļź╝ ļČäĒĢĀĒĢśļŖö ļŹ░ņŚÉļÅä ĒÜ©Ļ│╝ņĀüņØ┤ļØ╝Ļ│Ā ļ│┤Ļ│ĀĒĢśņśĆļŗż. Li ļō▒[49]ņØĆ MRI ņØ┤ļ»Ėņ¦Ćļź╝ CNN ļ¬©ļŹĖņŚÉ ĒĢÖņŖĄņŗ£ņ╝£ ļ╣äņØĖļæÉņĢöņØś ļ│æļ│ĆņØä ņ×ÉļÅÖ ļČäĒĢĀĒĢśļŖö ļ¬©ļŹĖņØä ļ¦īļōżņŚłĻ│Ā, ņØ┤ ļ¬©ļŹĖņØś dice similarity coefficient(DSC)ļŖö 0.89┬▒0.05ļĪ£ ļåÆņØĆ ņĀĢĒÖĢņä▒ņØä ļ│┤Ļ│ĀĒĢśņśĆļŗż. Liang ļō▒[50]ļÅä ļ╣äņØĖļæÉņĢö ĒÖśņ×ÉņŚÉņä£ ļ░®ņé¼ņäĀ ņ╣śļŻī ļ▓öņ£äļź╝ Ļ▓░ņĀĢĒĢśĻĖ░ ņ£äĒĢ┤ ļæÉĻ▓ĮļČĆ CTņŚÉņä£ ņŻ╝ņÜö ņןĻĖ░ļź╝ ņ×ÉļÅÖ ļČäĒĢĀĒĢśļŖö CNN ļ¬©ļŹĖņØä ļ│┤Ļ│ĀĒĢśņśĆļŖöļŹ░, ļćīĻ░ä, ņĢłĻĄ¼, ņłśņĀĢņ▓┤, ĒøäļæÉ, ĻĄ¼Ļ░Ģ, ņ▓Öņłś, ņØ┤ĒĢśņäĀ, ĒĢśņĢģ, ņ£Āņ¢æļÅīĻĖ░ ļō▒ ļīĆļČĆļČäņØś ņןĻĖ░ņŚÉņä£ DSC 0.85 ņØ┤ņāüņØś ņĀĢĒÖĢņä▒ņØä ļ│┤ņśĆļŗż.
ņĄ£ĻĘ╝ņŚÉļŖö ļ╣äĻ│╝ ņśüņŚŁņØś ļ│æļ”¼ ļČäņĢ╝ņŚÉņä£ļÅä ļöźļ¤¼ļŗØ ĻĖ░ļ▓ĢņØä ĒÖ£ņÜ®ĒĢ£ ņŗ£ļÅäĻ░Ć ņ׳ņŚłļŗż. Dimauro ļō▒[51]ņØĆ ļööņ¦ĆĒäĖĒÖöļÉ£ nasal cytology ņØ┤ļ»Ėņ¦ĆņŚÉņä£ CNN ļ¬©ļŹĖņØä ņØ┤ņÜ®ĒĢśņŚ¼ ņäĖĒżļź╝ ņ×ÉļÅÖ ļČäļ”¼ĒĢ£ ļŗżņØī, ņäĖĒżļōżņØś ņóģļźśĻ╣īņ¦Ć ņ×ÉļÅÖņ£╝ļĪ£ ĻĄ¼ļČäĒĢśļŖö ņŚ░ĻĄ¼ļź╝ ņŗ£Ē¢ēĒĢśņśĆļŖöļŹ░, ņäĖĒżļź╝ ļČäļ”¼ĒĢśļŖöļŹ░ 97% ļ»╝Ļ░ÉļÅä, ņäĖĒż ņóģļźś ņ¦äļŗ©ņŚÉļŖö 99% ņĀĢļÅäņØś ņĀĢĒÖĢņä▒ņØä ļ│┤ņśĆļŗż.
ņØ┤ ļ░¢ņŚÉ ņĢłļ®┤ ņä▒ĒśĢ ļČäņĢ╝ņŚÉņä£ļÅä ļ©ĖņŗĀļ¤¼ļŗØ ĻĖ░ļ▓ĢļōżņØ┤ ņĀüņÜ®ļÉśĻ│Ā ņ׳ļŗż. Borsting ļō▒[52]ņØĆ ņØ╝ļ░śņØĖņŚÉņä£ ņĮö ņä▒ĒśĢ ņŗ£Ē¢ē ņŚ¼ļČĆļź╝ ĒīÉļŗ©ĒĢśĻĖ░ ņ£äĒĢ┤ 22686ņןņØś ņĮö ņä▒ĒśĢ ņĀäĒøä ņĢłļ®┤ ņé¼ņ¦äņØä CNN ļ¬©ļŹĖņŚÉņä£ ĒĢÖņŖĄņŗ£ņ╝░Ļ│Ā, ņĢłļ®┤ ņé¼ņ¦äļ¦īņ£╝ļĪ£ 85%ņŚÉņä£ ņĮö ņä▒ĒśĢ ņŚ¼ļČĆļź╝ ņĀĢĒÖĢĒ׳ ņśłņĖĪĒĢĀ ņłś ņ׳ņŚłļŗż. Dorfman ļō▒[53]ņØĆ ņĢłļ®┤ ņé¼ņ¦äņØä ĒåĄĒĢ┤ ļéśņØ┤ļź╝ ņśłņĖĪĒĢśļŖö CNN ņĢīĻ│Āļ”¼ņ”ś(Microsoft Azure Face API)ņŚÉ 100ļ¬ģņØś ņĮö ņä▒ĒśĢ ĒÖśņ×ÉļōżņØś ņłśņłĀ ņĀäĒøä ņé¼ņ¦äņØä ņĀäņØ┤ ĒĢÖņŖĄņŗ£Ēé© Ļ▓░Ļ│╝, ņĮö ņä▒ĒśĢ ņłśņłĀ Ēøä ĒÅēĻĘĀ ņĢĮ 3.1ļģä ņĀĢļÅä ņŗżņĀ£ ļéśņØ┤ļ│┤ļŗż ņĀŖĻ▓ī ļ│┤ņØĖļŗżĻ│Ā ļ│┤Ļ│ĀĒĢśĻĖ░ļÅä ĒĢśņśĆļŗż.
Ļ▓░ ļĪĀņØśļŻī ļČäņĢ╝ņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØ┤ļéś ļöźļ¤¼ļŗØĻ│╝ Ļ░ÖņØĆ ņØĖĻ│Ąņ¦ĆļŖźņØś ĒÖ£ņÜ®ņØĆ ņØ┤ņĀ£ Ēü░ ĒØÉļ”äņØ┤ ļÉśņŚłļŗż. ļ╣ģļŹ░ņØ┤Ēä░ņÖĆ ņĀĢļ░Ć ņØśĒĢÖ ņŗ£ļīĆņŚÉ ļ©ĖņŗĀļ¤¼ļŗØņØś ĒÖ£ņÜ®ņØĆ ņĀÉņ░© ļŖśņ¢┤ļéĀ Ļ▓āņØ┤Ļ│Ā, ļ©ĖņŗĀļ¤¼ļŗØ ņĢīĻ│Āļ”¼ņ”śņØś Ļ░£ļ░£ņŚÉņä£ Ļ░Ćņן ņżæņÜöĒĢ£ ļČĆļČäņØĖ ņ¢æņ¦łņØś ļŹ░ņØ┤Ēä░ļź╝ ņČ®ļČäĒ׳ ņĀ£Ļ│ĄĒĢśĻĖ░ ņ£äĒĢ┤ņä£ ņ×äņāü ņØśņé¼ļōżņØś ņŚŁĒĢĀļÅä ņĀÉņ░© ņ╗żņ¦ł Ļ▓āņØ┤ļŗż. ņØ┤ļ╣äņØĖĒøäĻ│╝ ņØśņé¼ ņŚŁņŗ£ ņ×äņāüņŚÉņä£ ļ©ĖņŗĀļ¤¼ļŗØņØä ĒÖ£ņÜ®ĒĢśĻĖ░ ņ£äĒĢ┤ņä£ļŖö ļ©ĖņŗĀļ¤¼ļŗØ ņĢīĻ│Āļ”¼ņ”śņØś ĻĖ░ļ│Ė Ļ░£ļģÉņØä ņĢīĻ│Ā ļŹ░ņØ┤Ēä░ Ļ│╝ĒĢÖņ×ÉļōżĻ│╝ ņåīĒåĄĒĢĀ ņłś ņ׳ļŖö ļŖźļĀźņØä ĻĖĖļ¤¼ņĢ╝ ĒĢĀ Ļ▓āņØ┤ļŗż.
Fig.┬Ā1.Relationship between AI, machine learning, and deep learning. AI: artificial intelligence, KNN: k-nearest neighbors, SVM: support vector machine, ANN: artificial neural network, MLP: multiple layer perceptron, CNN: convolutional neural network, RNN: recurrent neural network. 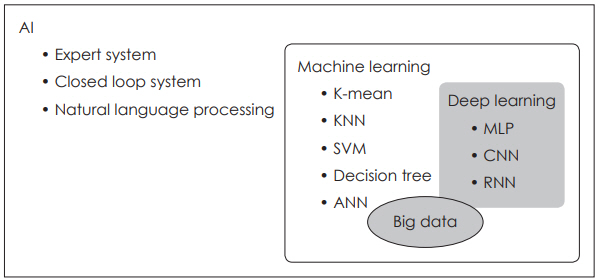
Fig.┬Ā2.Commonly used machine learning algorithms. KNN: k-nearest neighbors, SVM: support vector machine, MLP: multiple layer perceptron, CNN: convolutional neural network, RNN: recurrent neural network, DQN: deep Q network. 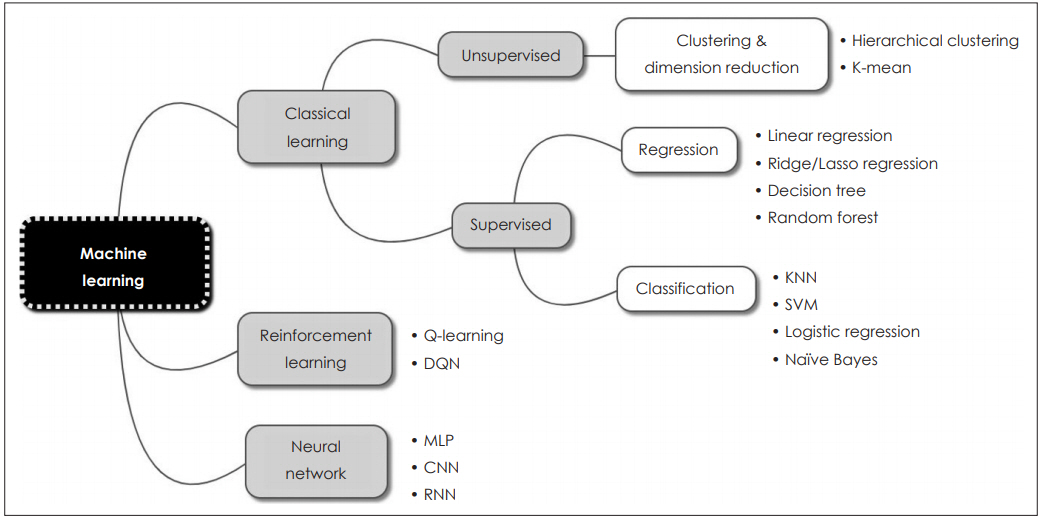
Fig.┬Ā4.A conceptual explanation of CNN. CNN consists of a number of convolutional and pooling layers, and then followed by one or more fully connected layers as in a standard multilayer neural network. CNN: convolutional neural network. 
Table┬Ā1.The differences between deep learning and machine learning Table┬Ā2.Summary of studies with machine learning application in rhinology
REFERENCES1. Bur AM, Shew M, New J. Ar tif icial intelligence for the otolaryngologist: A state of the art review. Otolaryngol Head Neck Surg 2019;160(4):603-11.
2. Bzdok D, Altman N, Krzywinski M. Statistics versus machine learning. Nat Methods 2018;15(4):233-4.
3. McCulloch WS, Pitts W. A logical calculus of the ideas immanent in nervous activity. Bull Math Biophys 1943;5(4):115-33.
4. Rosenblatt F. The perceptron: A probabilistic model for information storage and organization in the brain. Psychol Rev 1958;65(6):386-408.
5. Griewank A. Who invented the reverse mode of differentiation? Doc Math Extra Vol. Optimization Stories 2012;389-400.
6. Nair V, Hinton GE. Rectified linear units improve restricted boltzmann machines. ICML 2010.
7. Dahl GE, Sainath TN, Hinton GE. Improving deep neural networks for LVCSR using rectified linear units and dropout. Proceedings of the 2013 IEEE International Conference on Acoustics, Speech and Signal Processing;2013 May 26-31, Vancouver, Canada.
8. Ehteshami Bejnordi B, Veta M, Johannes van Diest P, van Ginneken B, Karssemeijer N, Litjens G, et al. Diagnostic assessment of deep learning algorithms for detection of lymph node metastases in women with breast cancer. JAMA 2017;318(22):2199-210.
9. Gulshan V, Peng L, Coram M, Stumpe MC, Wu D, Narayanaswamy A, et al. Development and validation of a deep learning algorithm for detection of diabetic retinopathy in retinal fundus photographs. JAMA 2016;316(22):2402-10.
10. Esteva A, Kuprel B, Novoa RA, Ko J, Swetter SM, Blau HM, et al. Dermatologist-level classification of skin cancer with deep neural networks. Nature 2017;542(7639):115-8.
12. Lee HC, Ryu HG, Chung EJ, Jung CW. Prediction of bispectral index during target-controlled infusion of propofol and remifentanil: A deep learning approach. Anesthesiology 2018;128(3):492-501.
13. Park SH. Artificial intelligence in medicine: BeginnerŌĆÖs guide. J Korean Soc Radiol 2018;78(5):301-8.
14. van der Veen J, Willems S, Deschuymer S, Robben D, Crijns W, Maes F, et al. Benefits of deep learning for delineation of organs at risk in head and neck cancer. Radiother Oncol 2019;138:68-74.
15. Huang B, Chen Z, Wu PM, Ye Y, Feng ST, Wong CO, et al. Fully automated delineation of gross tumor volume for head and neck cancer on PET-CT using deep learning: A dual-center study. Contrast Media Mol Imaging 2018;2018:8923028.
16. Ren J, Jing X, Wang J, Ren X, Xu Y, Yang Q, et al. Automatic recognition of laryngoscopic images using a deep-learning technique. Laryngoscope 2020.
17. Mascharak S, Baird BJ, Holsinger FC. Detecting oropharyngeal carcinoma using multispectral, narrow-band imaging and machine learning. Laryngoscope 2018;128(11):2514-20.
18. Tamashiro A, Yoshio T, Ishiyama A, Tsuchida T, Hijikata K, Yoshimizu S, et al. Artificial intelligence-based detection of pharyngeal cancer using convolutional neural networks. Dig Endosc 2020.
19. Xiong H, Lin P, Yu JG, Ye J, Xiao L, Tao Y, et al. Computer-aided diagnosis of laryngeal cancer via deep learning based on laryngoscopic images. EBioMedicine 2019;48:92-9.
20. Halicek M, Little JV, Wang X, Chen AY, Fei B. Optical biopsy of head and neck cancer using hyperspectral imaging and convolutional neural networks. J Biomed Opt 2019;24(3):1-9.
21. Karadaghy OA, Shew M, New J, Bur AM. Development and assessment of a machine learning model to help predict survival among patients with oral squamous cell carcinoma. JAMA Otolaryngol Head Neck Surg 2019;145(12):1115-20.
22. Formeister EJ, Baum R, Knott PD, Seth R, Ha P, Ryan W, et al. Machine learning for predicting complications in head and neck microvascular free tissue transfer. Laryngoscope 2020.
23. Fujima N, Shimizu Y, Yoshida D, Kano S, Mizumachi T, Homma A, et al. Machine-learning-based prediction of treatment outcomes using MR imaging-derived quantitative tumor information in patients with sinonasal squamous cell carcinomas: A preliminary study. Cancers (Basel) 2019;11(6):800.
24. Mermod M, Jourdan EF, Gupta R, Bongiovanni M, Tolstonog G, Simon C, et al. Development and validation of a multivariable prediction model for the identification of occult lymph node metastasis in oral squamous cell carcinoma. Head Neck 2020;42(8):1811-20.
25. Alabi RO, Elmusrati M, Sawazaki-Calone I, Kowalski LP, Haglund C, Coletta RD, et al. Machine learning application for prediction of locoregional recurrences in early oral tongue cancer: A Web-based prognostic tool. Virchows Arch 2019;475(4):489-97.
26. Tighe D, Lewis-Morris T, Freitas A. Machine learning methods applied to audit of surgical outcomes after treatment for cancer of the head and neck. Br J Oral Maxillofac Surg 2019;57(8):771-7.
27. Shew M, New J, Bur AM. Machine learning to predict delays in adjuvant radiation following surgery for head and neck cancer. Otolaryngol Head Neck Surg 2019;160(6):1058-64.
28. Bur AM, Holcomb A, Goodwin S, Woodroof J, Karadaghy O, Shnayder Y, et al. Machine learning to predict occult nodal metastasis in early oral squamous cell carcinoma. Oral Oncol 2019;92:20-5.
29. Bing D, Ying J, Miao J, Lan L, Wang D, Zhao L, et al. Predicting the hearing outcome in sudden sensorineural hearing loss via machine learning models. Clin Otolaryngol 2018;43(3):868-74.
30. Park KV, Oh KH, Jeong YJ, Rhee J, Han MS, Han SW, et al. Machine learning models for predicting hearing prognosis in unilateral idiopathic sudden sensorineural hearing loss. Clin Exp Otorhinolaryngol 2020;13(2):148-56.
31. Cha D, Shin SH, Kim SH, Choi JY, Moon IS. Machine learning approach for prediction of hearing preservation in vestibular schwannoma surgery. Sci Rep 2020;10(1):7136.
32. Abouzari M, Goshtasbi K, Sarna B, Khosravi P, Reutershan T, Mostaghni N, et al. Prediction of vestibular schwannoma recurrence using artificial neural network. Laryngoscope Investig Otolaryngol 2020;5(2):278-85.
33. Kim H, Kang WS, Park HJ, Lee JY, Park JW, Kim Y, et al. Cochlear implantation in postlingually deaf adults is time-sensitive towards positive outcome: Prediction using advanced machine learning techniques. Sci Rep 2018;8(1):18004.
34. Livingstone D, Talai AS, Chau J, Forkert ND. Building an otoscopic screening prototype tool using deep learning. J Otolaryngol Head Neck Surg 2019;48(1):66.
35. Wang YM, Li Y, Cheng YS, He ZY, Yang JM, Xu JH, et al. Deep learning in automated region proposal and diagnosis of chronic otitis media based on computed tomography. Ear Hear 2020;41(3):669-77.
36. Hallac RR, Lee J, Pressler M, Seaward JR, Kane AA. Identifying ear abnormality from 2D photographs using convolutional neural networks. Sci Rep 2019;9(1):18198.
37. Livingstone D, Chau J. Otoscopic diagnosis using computer vision: An automated machine learning approach. Laryngoscope 2020;130(6):1408-13.
38. Habib AR, Wong E, Sacks R, Singh N. Artificial intelligence to detect tympanic membrane perforations. J Laryngol Otol 2020;134(4):311-5.
39. George-Jones NA, Wang K, Wang J, Hunter JB. Automated detection of vestibular schwannoma growth using a two-dimensional U-Net convolutional neural network. Laryngoscope; 2020.
40. Soler ZM, Hyer JM, Rudmik L, Ramakrishnan V, Smith TL, Schlosser RJ. Cluster analysis and prediction of treatment outcomes for chronic rhinosinusitis. J Allergy Clin Immunol 2016;137(4):1054-62.
41. Lal D, Hopkins C, Divekar RD. SNOT-22-based clusters in chronic rhinosinusitis without nasal polyposis exhibit distinct endotypic and prognostic differences. Int Forum Allergy Rhinol 2018;8(7):797-805.
42. Tomassen P, Vandeplas G, Van Zele T, Cardell LO, Arebro J, Olze H, et al. Inflammatory endotypes of chronic rhinosinusitis based on cluster analysis of biomarkers. J Allergy Clin Immunol 2016;137(5):1449-56, e4.
43. Kim JW, Huh G, Rhee CS, Lee CH, Lee J, Chung JH, et al. Unsupervised cluster analysis of chronic rhinosinusitis with nasal polyp using routinely available clinical markers and its implication in treatment outcomes. Int Forum Allergy Rhinol 2019;9(1):79-86.
44. Walker A, Surda P. Unsupervised learning techniques for the investigation of chronic rhinosinusitis. Ann Otol Rhinol Laryngol 2019;128(12):1170-6.
45. Chowdhury NI, Smith TL, Chandra RK, Turner JH. Automated classification of osteomeatal complex inflammation on computed tomography using convolutional neural networks. Int Forum Allergy Rhinol 2019;9(1):46-52.
46. Huang J, Habib AR, Mendis D, Chong J, Smith M, Duvnjak M, et al. An artificial intelligence algorithm that differentiates anterior ethmoidal artery location on sinus computed tomography scans. J Laryngol Otol 2020;134(1):52-5.
47. Humphries SM, Centeno JP, Notary AM, Gerow J, Cicchetti G, Katial RK, et al. Volumetric assessment of paranasal sinus opacification on computed tomography can be automated using a convolutional neural network. Int Forum Allergy Rhinol; 2020.
48. Li C, Jing B, Ke L, Li B, Xia W, He C, et al. Development and validation of an endoscopic images-based deep learning model for detection with nasopharyngeal malignancies. Cancer Commun (Lond) 2018;38(1):59.
49. Li Q, Xu Y, Chen Z, Liu D, Feng ST, Law M, et al. Tumor segmentation in contrast-enhanced magnetic resonance imaging for nasopharyngeal carcinoma: Deep learning with convolutional neural network. Biomed Res Int 2018;2018:9128527.
50. Liang S, Tang F, Huang X, Yang K, Zhong T, Hu R, et al. Deeplearning-based detection and segmentation of organs at risk in nasopharyngeal carcinoma computed tomographic images for radiotherapy planning. Eur Radiol 2019;29(4):1961-7.
51. Dimauro G, Ciprandi G, Deperte F, Girardi F, Ladisa E, Latrofa S, et al. Nasal cytology with deep learning techniques. Int J Med Inform 2019;122:13-9.
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||

 |
 |